Der DNA-Fingerabdruck
Wie die Fingerabdrücke, die in den 1930er Jahren von Detektiven und Polizeilaboren in Gebrauch genommen wurden, hat jeder Mensch einen einzigartigen DNA-Fingerabdruck. Im Gegensatz zu einem herkömmlichen Fingerabdruck, der nur an den Fingerspitzen vorkommt und durch eine Operation verändert werden kann, ist der DNA-Fingerabdruck bei jeder Zelle, jedem Gewebe und jedem Organ eines Menschen gleich. Er kann durch keine bekannte Behandlung verändert werden. Daher wird der DNA-Fingerabdruck immer mehr zur wichtigsten Methode zur Identifizierung und Unterscheidung einzelner Menschen. DNA-Fingerabdrücke sind auch in verschiedenen Anwendungen der Gesundheitsforschung und bei der Diagnose von Erbkrankheiten nützlich. DNA-Fingerabdrücke werden oft in der Justiz verwendet, um Verdächtige mit biologischen Beweisen in Verbindung zu bringen und Vaterschaftsfälle zu lösen.
Wie wird der DNA-Fingerabdruck verwendet, um einen Verbrecher zu identifizieren?
Oxford Education (YouTube)
Die Polymerase-Kettenreaktion
Die PCR bereitete den Boden für eine wissenschaftliche Revolution
Im Jahr 1983 entwickelte Kary Mullis von der Cetus Corporation eine neue Technik, die Polymerase-Kettenreaktion (PCR). Das Ziel der PCR ist es, eine große Menge an DNA in einem Reagenzglas (in vitro) herzustellen, ausgehend von einer geringen Menge. Technisch gesehen bedeutet dies die kontrollierte Replikation einer DNA-Sequenz oder eines Gens von Interesse. Die Template-Stränge können jede Form von doppelsträngiger DNA sein, wie z. B. genomische DNA. Ein Forscher kann Spuren von genomischer DNA aus einem Blutstropfen, einem einzelnen Haarfollikel oder einer Wangenzelle entnehmen (theoretisch wird nur ein einziger Vorlagenstrang benötigt, um Millionen neuer identischer DNA-Moleküle zu kopieren und zu erzeugen) und damit genug für eine Untersuchung herstellen. Vor der PCR wäre dies unmöglich gewesen.
PCR-Amplifikation
Es wird geschätzt, dass es 30.000-50.000 einzelne Gene im menschlichen Genom gibt. Die wahre Stärke der PCR ist die Fähigkeit, ein bestimmtes Stück DNA (oder ein Gen) aus einem kompletten Genom anzuvisieren und Millionen von Kopien davon herzustellen (oder zu amplifizieren).
Das Rezept für eine PCR-Amplifikation von DNA enthält eine einfache Mischung von Zutaten, die als die Stücke erkennbar sein sollten, die zur Replikation von DNA im Körper verwendet werden:
- DNA-Template – enthält die intakte Sequenz der zu amplifizierenden DNA
- Einzelne Desoxynukleotide (A, T, G, und C) – Rohmaterial der DNA
- DNA-Polymerase – ein Enzym, das die Nukleotide zu einer neuen DNA-Kette zusammensetzt
- Magnesiumionen – ein Cofaktor (Katalysator), der von der DNA-Polymerase zur Bildung der DNA-Kette benötigt wird
- Oligonukleotid-Primer – DNA-Stücke, die komplementär zur Matrize sind und der DNA-Polymerase genau sagen, wo sie mit dem Kopieren beginnen soll
- Salzpuffer – sorgt für die optimale ionische Umgebung und den optimalen pH-Wert für die PCR-Reaktion
Die Matrizen-DNA kann aus Blut, Haut, Sperma oder Haaren etc. stammen. Wenn alle anderen Komponenten unter den richtigen Bedingungen kombiniert werden, entsteht eine Kopie des ursprünglichen doppelsträngigen Template-DNA-Moleküls – eine Verdopplung der Anzahl der Template-Stränge. Jedes Mal, wenn dieser Zyklus wiederholt wird, werden Kopien von Kopien gemacht und die Anzahl der Template-Stränge verdoppelt sich – von 2 auf 4 auf 8 auf 16 und so weiter – bis nach 20 Zyklen 1.048.554 exakte Kopien der Zielsequenz vorliegen.
PCR macht sich dieselben grundlegenden Prozesse zunutze, die Zellen zur Vervielfältigung ihrer DNA verwenden.
- Komplementäre DNA-Strang-Hybridisierung
- DNA-Strang-Synthese durch DNA-Polymerase
Die DNA-Primer sind so konzipiert, dass sie eine DNA-Sequenz innerhalb des Genoms flankieren und somit das exakte Startsignal für die DNA-Polymerase liefern, damit diese bindet und mit der Synthese (Replikation) von Kopien dieser Ziel-DNA beginnt. Die Komplementärstrang-Hybridisierung findet statt, wenn verschiedene Primer an die jeweiligen komplementären Basensequenzen auf der Template-DNA annealen oder binden. Die Primer sind kurze einzelsträngige DNA-Moleküle (~20 Basen lang), von denen einer komplementär zu einem Teil des 5′-3′-Strangs und ein anderer komplementär zu einem Teil des 3′-5′-Strangs der Vorlage ist. Diese Primer lagern sich an die getrennten Template-Stränge an und dienen als Startpunkte für die DNA-Taq-Replikation durch die DNA-Polymerase.
Taq-DNA-Polymerase verlängert die gelagerten Primer, indem sie den Template-Strang „liest“ und die komplementäre Sequenz synthetisiert. Auf diese Weise repliziert die Taq-Polymerase die beiden Template-DNA-Stränge. Diese Polymerase wurde aus einem hitzestabilen Bakterium (Thermus Aquaticus) isoliert, das in der Natur in den Dampfschloten im Yellowstone National Park lebt. Aus diesem Grund haben sich die Enzyme innerhalb dieser Bakterien so entwickelt, dass sie hohen Temperaturen (94°C) standhalten und in der PCR-Reaktion verwendet werden können.
PCR Schritt für Schritt
Die PCR-Amplifikation umfasst drei Hauptschritte, einen Denaturierungsschritt, einen Annealing-Schritt und einen Verlängerungsschritt (zusammengefasst in der folgenden Abbildung).
1) Denaturierung: Bei der Denaturierung wird das Reaktionsgemisch für 1 Minute auf 94°C erhitzt, was zum Schmelzen bzw. zur Trennung des doppelsträngigen DNA-Templates in zwei einzelsträngige Moleküle führt.
2) Amplifikation: Bei der PCR-Amplifikation muss die DNA-Vorlage aufgetrennt werden, bevor die Polymerase eine neue Kopie erzeugen kann. Die hohe Temperatur, die erforderlich ist, um die DNA-Stränge zu schmelzen, würde normalerweise die Aktivität der meisten Enzyme zerstören, aber da die Taq-Polymerase aus Bakterien isoliert wurde, die in den hohen Temperaturen von heißen Quellen gedeihen, bleibt sie aktiv.
3) Annealing: Während des Annealing-Schrittes „annealen“ die Oligonukleotid-Primer an oder finden ihre komplementären Sequenzen auf den beiden einzelsträngigen Template-Strängen der DNA. In diesen annealierten Positionen können sie als Primer für die Taq-DNA-Polymerase fungieren. Sie werden Primer genannt, weil sie die Synthese eines neuen Strangs „vorbereiten“, indem sie eine kurze Sequenz doppelsträngiger DNA für die Taq-Polymerase bereitstellen, von der aus sie sich verlängern und einen neuen komplementären Strang aufbauen kann. Die Bindung der Primer an ihre Template-Sequenzen ist ebenfalls stark temperaturabhängig.
Während des Verlängerungsschritts besteht die Aufgabe der Taq-DNA-Polymerase darin, die Nukleotide (A, T, G und C) nacheinander an den Primer anzuhängen, um eine komplementäre Kopie der DNA-Vorlage zu erstellen.

Während der Polymerisation beträgt die Reaktionstemperatur 72°C, die Temperatur, die eine optimale Taq-Polymerase-Aktivität erzeugt. Die drei Schritte Denaturierung, Extension und Annealing bilden einen „Zyklus“ der PCR. Eine komplette PCR-Amplifikation durchläuft 40 Zyklen.
Die gesamte 40-Zyklen-Reaktion wird in einem Reagenzglas durchgeführt, das in einen Thermocycler gestellt wird. Der Thermocycler enthält einen Aluminiumblock, der die Proben aufnimmt und über große Temperaturunterschiede schnell aufgeheizt und abgekühlt werden kann. Das schnelle Aufheizen und Abkühlen dieses Thermoblocks wird als Temperaturzyklus oder Thermocycler bezeichnet.
PCR-Temperaturzyklus = Denaturierungsschritt (94°C) + Annealingschritt (60°C) + Verlängerungsschritt (72°C)
DNA Fingerprinting
Variable Number of Tandem Repeats (VNTR)
Das evolutionäre Prinzip der Variation innerhalb einer Population ist ein Eckpfeiler der Biologie. Diese Variation resultiert aus subtilen Unterschieden in der DNA-Sequenz bei Individuen einer bestimmten Art. Ein Ursprung der Variation ergibt sich, wenn es während der DNA-Replikation zur Duplikation einer kleinen Nukleotidsequenz kommt. Dies führt zu einer Tandemwiederholung der ursprünglichen Sequenz. Wenn dieser Fehler bei einer weiteren Replikationsrunde erneut auftritt, liegen drei Kopien einer Sequenz im Tandem vor, wie im Folgenden dargestellt:
Individuum 1: GTACTCCAATCATGTACCATGAC
Individuum 2: GTACTCCAATCATCATGTACCATGAC
Individuum 3: GTACTCCAATCATCATCATCATGTACCATGAC
Hier sind einzelne DNA-Stränge vom gleichen Locus und drei verschiedenen Individuen dargestellt. CAT‘ wird ein-, zwei- oder dreimal wiederholt, was zu unterschiedlich langen Allelen führt.
Es gibt zwei Standardmethoden für den DNA-Fingerabdruck:
- PCR von DNA, die VNTRs enthält.
- Southern Blotting (mit RFLPs).
PCR ermöglicht die Vervielfältigung einer einzelnen DNA-Kopie in Millionen von Kopien. Diese Technik erfordert jedoch, dass die DNA von Interesse bereits eine bekannte Sequenz aufweist, um Primer zu entwerfen, die spezifisch mit der Ziel-DNA hybridisieren. Eine solche Region, die in mehreren Ländern für die forensische Analyse von DNA-Proben verwendet wird, ist der D1S80-Lokus. Der D1S80-Locus befindet sich im distalen Teil des kurzen Arms von Chromosom 1 und enthält eine variable Anzahl von Tandem-Repeats.

D1S80 Beispiel Tandem-Repeats
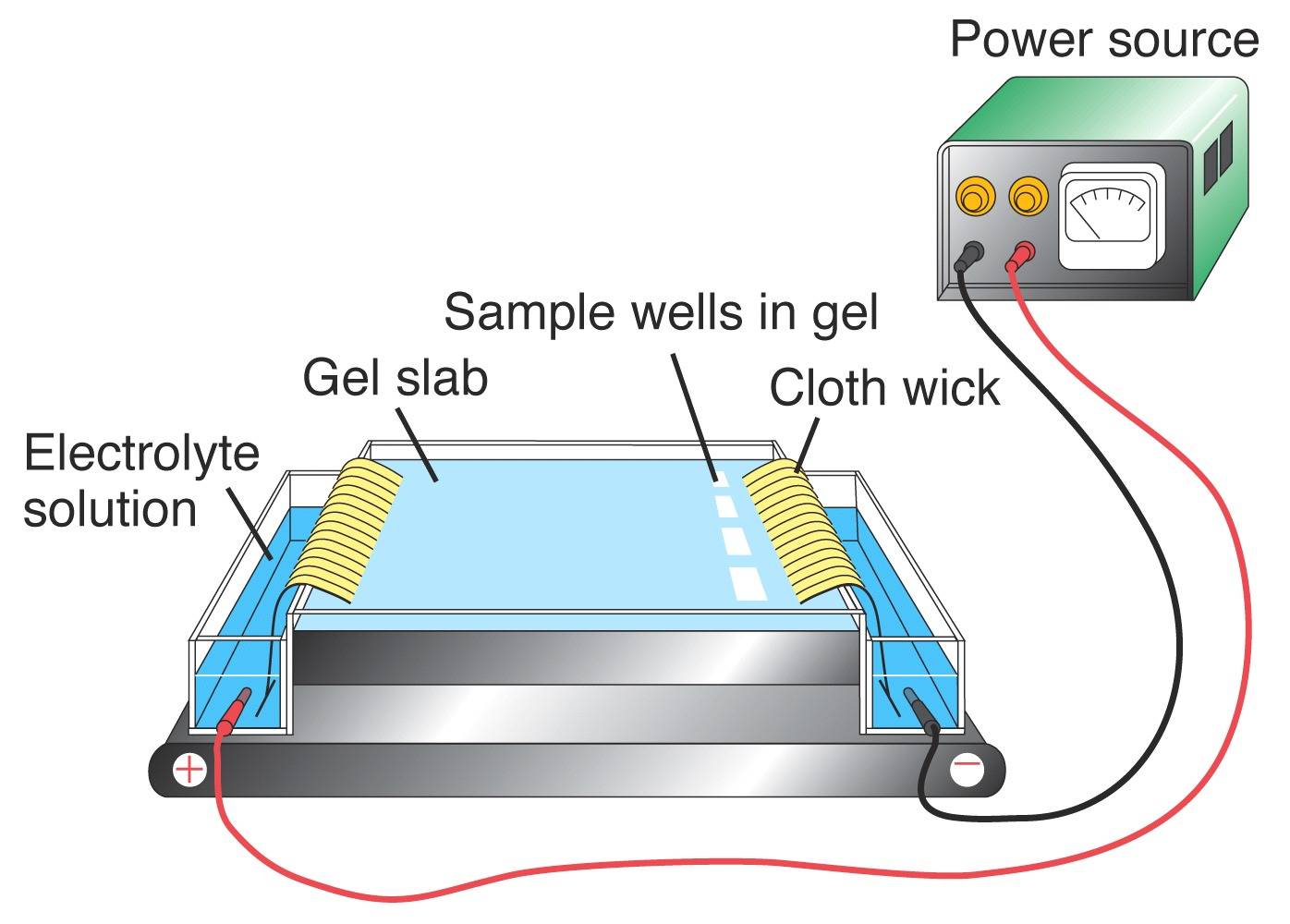
Elektrophorese
Mit Hilfe der PCR können DNA-Abschnitte amplifiziert werden, von denen bekannt ist, dass sie VNTRs enthalten. Das resultierende Produkt wird dann betrachtet, um den DNA-Genotyp der Person zu erhalten. Um das DNA-Profil von einem bestimmten Locus sichtbar zu machen, müssen die DNA-Fragmente mit Hilfe der Gelelektrophorese nach ihrer Größe aufgetrennt werden. Ein elektrischer Strom wird an eine Agarosegelmatrix angelegt. Da die DNA aufgrund der Phosphatgruppen negativ geladen ist, bewegen sich die Fragmente in Richtung des positiven Pols. DNA-Moleküle unterschiedlicher Größe wandern mit unterschiedlicher Geschwindigkeit durch das Gel, wobei sich die kleineren Fragmente schneller bewegen als die größeren. Sobald die DNA im Gel gefärbt ist, kann sie visualisiert werden, um das DNA-Profil zu erkennen.

(Links) Fiktives Agarosegel. Die Lanes 1, 2 und 3 repräsentieren Banden mit den Daten der oben genannten Personen. Lane 4 repräsentiert DNA von einem Tatort. (Rechts) Typische Agarosegel-Apparatur.
Ethidiumbromid (EtBr) ist ein großes, flaches Basismolekül, das einem DNA-Basenpaar ähnelt.

Ethidiumbromid
Aufgrund seiner chemischen Struktur kann es in einen DNA-Strang interkalieren (oder einfügen). Ethidiumbromid wird in molekularbiologischen Labors häufig zum Färben von Elektrophoresegelen verwendet. Die Verbindung bildet fluoreszierende Komplexe mit Nukleinsäuren und diese können unter UV-Licht betrachtet werden. EtBr wird der erwärmten Agarose vor dem Gießen des Gels zugesetzt, um eine Endkonzentration von 0,5ug/ml zu erhalten. EtBr ist ein Mutagen und muss mit äußerster Vorsicht und mit behandschuhten Händen gehandhabt werden.
Sequenzierung – Hintergrund
RFLP (oft „rif lip“ ausgesprochen, als ob es ein Wort wäre) ist eine Methode, die von Molekularbiologen verwendet wird, um eine bestimmte Sequenz von DNA zu verfolgen, während sie an andere Zellen weitergegeben wird. RFLPs können in vielen verschiedenen Bereichen eingesetzt werden, um unterschiedliche Ziele zu erreichen. RFLPs können in Vaterschaftsfällen oder Kriminalfällen verwendet werden, um die Quelle einer DNA-Probe zu bestimmen. RFLPs können auch verwendet werden, um den Krankheitsstatus eines Individuums zu bestimmen.
Jeder Organismus erbt seine DNA von seinen Eltern. Da die DNA mit jeder Generation repliziert wird, kann jede gegebene Sequenz an die nächste Generation weitergegeben werden. Ein RFLP ist eine Sequenz von DNA, die an jedem Ende eine Restriktionsstelle mit einer „Ziel“-Sequenz dazwischen hat. Eine Zielsequenz ist ein beliebiges Segment der DNA, das an eine Sonde binden kann, indem es komplementäre Basenpaare bildet. Eine Sonde ist eine Sequenz von einzelsträngiger DNA, die mit Radioaktivität oder einem Enzym markiert wurde, so dass die Sonde nachgewiesen werden kann. Wenn eine Sonde ein Basenpaar mit ihrem Ziel bildet, kann der Untersucher diese Bindung nachweisen und weiß, wo sich die Zielsequenz befindet, da die Sonde nachweisbar ist. RFLP erzeugt eine Reihe von Banden, wenn ein Southern Blot mit einer bestimmten Kombination aus Restriktionsenzym und Sondensequenz durchgeführt wird.
Beispielsweise wollen wir eine bestimmte RFLP verfolgen, die durch das Restriktionsenzym EcoR I und die Zielsequenz von 20 Basen GCATGCATGCATGCATGCAT definiert ist. EcoR I bindet an seine Erkennungssequenz GAATTC und schneidet die doppelsträngige DNA wie gezeigt:

In dem unten gezeigten DNA-Segment können Sie die Elemente einer RFLP sehen; eine Zielsequenz, die von einem Paar Restriktionsstellen flankiert wird. Wenn dieses DNA-Segment mit EcoR I geschnitten wird, entstehen drei Restriktionsfragmente, aber nur eines enthält die Zielsequenz, die von der komplementären Sondensequenz (lila) gebunden werden kann.

Betrachten wir zwei Personen und die DNA-Segmente, die sie tragen und die diesen RFLP enthalten (aus Gründen der Übersichtlichkeit sehen wir nur einen der beiden DNA-Stände). Da Jack und Jill beide diploide (doppelsträngige DNA) Organismen sind, haben sie zwei Kopien dieses RFLP. Wenn wir eine Kopie von Jack und eine Kopie von Jill untersuchen, sehen wir, dass sie identisch sind:
- Jack 1: -GAATTC—(8.2 kb)—GCATGCATGCATGCATGCAT—(4.2 kb)—GAATTC-
- Jill 1: -GAATTC—(8.2 kb)—GCATGCATGCATGCAT—(4.2 kb)—GAATTC-
Wenn wir ihre zweiten Kopien dieses RFLP untersuchen, sehen wir, dass sie nicht identisch sind. Jack 2 fehlt eine EcoR I Restriktionsstelle, die Jill 1,2 kb stromaufwärts der Zielsequenz hat (Unterschied in Kursivschrift).
- Jack 2: -GAATTC–(1.8 kb)-CCCTTT–(1.2 kb)–GCATGCATGCATGCATGCAT–(1.3 kb)-GAATTC-
- Jill 2: -GAATTC–(1.8 kb)-GAATTC–(1.2 kb)–GCATGCATGCATGCATGCAT–(1.3 kb)-GAATTC-
Wenn also Jack und Jill ihre DNA einer RFLP-Analyse unterziehen, haben sie eine gemeinsame Bande und eine Bande, die im Molekulargewicht nicht mit der des anderen übereinstimmt:

Southern-Blot-Methode
Dies ist ein kurzer Überblick darüber, wie ein Southern-Blot (formaler als DNA-Blot bezeichnet) durchgeführt wird und welche Art von Daten man daraus erhalten kann. Das Ergebnis eines Southern Blots ist das, woran die meisten Leute denken, wenn sie von einem „DNA-Fingerabdruck“ sprechen.

Southern Blots ermöglichen es den Forschern, das Molekulargewicht eines Restriktionsfragments zu bestimmen und relative Mengen in verschiedenen Proben zu messen.
Vorgehensweise:
- DNA (genomische oder andere Quelle) wird mit einem Restriktionsenzym verdaut und durch Gelelektrophorese aufgetrennt, normalerweise ein Agarosegel. Da es so viele verschiedene Restriktionsfragmente auf dem Gel gibt, erscheint es in der Regel als Schlieren und nicht als diskrete Banden. Die DNA wird durch Inkubation mit NaOH zu Einzelsträngen denaturiert.
- Die DNA wird auf eine Membran übertragen, die aus einem Blatt speziellen Blotting-Papiers besteht. Die DNA-Fragmente behalten das gleiche Trennungsmuster bei, das sie auf dem Gel hatten.
- Der Blot wird mit vielen Kopien einer Sonde inkubiert, die aus einzelsträngiger DNA besteht. Diese Sonde bildet Basenpaare mit ihrer komplementären DNA-Sequenz und bindet, um ein doppelsträngiges DNA-Molekül zu bilden. Die Sonde kann nicht gesehen werden, aber sie ist entweder radioaktiv oder hat ein Enzym gebunden (z. B. alkalische Phosphatase oder Meerrettichperoxidase).
- Die Position der Sonde wird durch Inkubation mit einem farblosen Substrat offenbart, das das gebundene Enzym in ein farbiges Produkt umwandelt, das gesehen werden kann oder Licht abgibt, das einen Röntgenfilm belichtet. Wenn die Sonde mit Radioaktivität markiert wurde, kann sie den Röntgenfilm direkt belichten.
Unten sehen Sie ein Beispiel für einen echten Southern Blot, der verwendet wurde, um das Vorhandensein eines Gens nachzuweisen, das in eine gemischte Zellpopulation transformiert wurde. In diesem Southern Blot lässt sich leicht feststellen, welche Zellen das Gen inkorporiert haben und welche nicht.

Die Abbildung links zeigt ein Foto eines 0,7%igen Agarosegels, auf das 14 verschiedene Proben geladen wurden (plus Molekulargewichtsmarker in der ganz rechten Spur und ein leuchtendes Lineal, das zur Analyse der Ergebnisse verwendet wird). Jede DNA-Probe wurde mit demselben Restriktionsenzym (EcoRI) verdaut. Beachten Sie, dass die DNA nicht als eine Reihe von diskreten Banden erscheint, sondern eher als ein Schlieren. Die Abbildung rechts ist eine Kopie des Röntgenfilms und zeigt, welche Stämme die Ziel-DNA enthalten und welche nicht.
Ein Laser-Scan des Gels erzeugt einen genomischen Fingerabdruck der folgenden Art:
