La huella dactilar de ADN
Al igual que las huellas dactilares que empezaron a utilizar los detectives y los laboratorios policiales durante la década de 1930, cada persona tiene una huella dactilar de ADN única. A diferencia de la huella dactilar convencional, que se produce sólo en las yemas de los dedos y puede ser alterada por la cirugía, la huella de ADN es la misma para cada célula, tejido y órgano de una persona. No puede ser alterada por ningún tratamiento conocido. Por consiguiente, la huella de ADN se está convirtiendo rápidamente en el principal método para identificar y distinguir a los seres humanos. Las huellas de ADN también son útiles en varias aplicaciones de la investigación sanitaria humana y para el diagnóstico de trastornos hereditarios. Las huellas de ADN se utilizan a menudo en el sistema judicial para relacionar a los sospechosos con las pruebas biológicas y resolver los casos de paternidad.
¿Cómo se utilizan las huellas de ADN para identificar a un criminal?
Oxford Education (YouTube)
La reacción en cadena de la polimerasa
La PCR sentó las bases de una revolución científica
En 1983, Kary Mullis, de Cetus Corporation, desarrolló una nueva técnica, denominada reacción en cadena de la polimerasa (PCR). El objetivo de la PCR es producir una gran cantidad de ADN en un tubo de ensayo (in vitro), partiendo de una cantidad mínima. Técnicamente hablando, esto significa la replicación controlada de una secuencia de ADN, o gen, de interés. La cadena molde puede ser cualquier forma de ADN de doble cadena, como el ADN genómico. Un investigador puede tomar cantidades mínimas de ADN genómico de una gota de sangre, de un solo folículo piloso o de una célula de la mejilla (en teoría, sólo se necesita una única hebra molde para copiar y generar millones de nuevas moléculas de ADN idénticas) y hacer lo suficiente para estudiar. Antes de la PCR, esto habría sido imposible.
Amplificación por PCR
Se calcula que hay entre 30.000 y 50.000 genes individuales en el genoma humano. El verdadero poder de la PCR es la capacidad de dirigir y hacer millones de copias de (o amplificar) un trozo específico de ADN (o gen) de un genoma completo.
La receta para una amplificación de ADN por PCR contiene una simple mezcla de ingredientes que debería ser reconocible como las piezas utilizadas para replicar el ADN en el cuerpo:
- Plantilla de ADN – que contiene la secuencia intacta de ADN que se va a amplificar
- Desoxinucleótidos individuales (A, T, G, y C) – materia prima del ADN
- Ana polimerasa – una enzima que ensambla los nucleótidos en una nueva cadena de ADN
- Iones de magnesio – un cofactor (catalizador) requerido por la ADN polimerasa para crear la cadena de ADN
- Primeros oligonucleótidos – trozos de ADN complementarios al molde que indican a la ADN polimerasa exactamente dónde empezar a hacer copias
- Tampón salino – proporciona el entorno iónico y el pH óptimos para la reacción de PCR
- Hibridación de la cadena de ADN complementaria
- Síntesis de la cadena de ADN a través de la ADN polimerasa
- PCR del ADN que contiene VNTRs.
- Southern blotting (utilizando RFLPs).
El ADN molde puede proceder de la sangre, piel, semen o cabello, etc. Cuando todos los demás componentes se combinan en las condiciones adecuadas, se hace una copia de la molécula original de ADN molde de doble cadena, duplicando el número de cadenas molde. Cada vez que se repite este ciclo, se hacen copias a partir de copias y el número de hebras plantilla se duplica -de 2 a 4 a 8 a 16 y así sucesivamente- hasta que después de 20 ciclos hay 1.048.554 copias exactas de la secuencia objetivo.
La PCR hace uso de los mismos procesos básicos que utilizan las células para duplicar su ADN.
Los cebadores de ADN están diseñados para flanquear una secuencia de ADN dentro del genoma y así proporcionar la señal de inicio exacta para que la ADN polimerasa se una y comience a sintetizar (replicar) copias de ese ADN objetivo. La hibridación de la cadena complementaria tiene lugar cuando diferentes cebadores se anejan, o se unen a cada una de sus respectivas secuencias de bases complementarias en el ADN molde. Los cebadores son moléculas cortas de ADN monocatenario (de unas 20 bases de longitud), una complementaria a una parte de la cadena 5′-3′ y otra complementaria a una parte de la cadena 3′-5′ del molde. Estos cebadores se acoplan a las hebras separadas del molde y sirven como puntos de partida para la replicación del ADN Taq por la ADN polimerasa.
La ADN polimerasa Taq extiende los cebadores recocidos «leyendo» la hebra del molde y sintetizando la secuencia complementaria. De este modo, la Taq polimerasa replica las dos cadenas de ADN molde. Esta polimerasa ha sido aislada de una bacteria termoestable (Thermus Aquaticus) que en la naturaleza vive dentro de los respiraderos de vapor del Parque Nacional de Yellowstone. Por esta razón, las enzimas dentro de estas bacterias han evolucionado para soportar altas temperaturas (94°C) y pueden ser utilizadas en la reacción de PCR.
La PCR paso a paso
La amplificación por PCR incluye tres pasos principales, un paso de desnaturalización, un paso de recocido y un paso de extensión (resumidos en la figura siguiente).
1) Desnaturalización: En la desnaturalización, la mezcla de reacción se calienta a 94°C durante 1 minuto, lo que provoca la fusión o separación del molde de ADN de doble cadena en dos moléculas de cadena simple.
2) Amplificación: En la amplificación por PCR, las plantillas de ADN deben separarse antes de que la polimerasa pueda generar una nueva copia. La alta temperatura necesaria para fundir las hebras de ADN normalmente destruiría la actividad de la mayoría de las enzimas, pero como la Taq polimerasa se aisló de bacterias que prosperan en las altas temperaturas de las aguas termales, permanece activa.
3) Recocido: Durante el paso de recocido, los cebadores de oligonucleótidos se «acoplan» o encuentran sus secuencias complementarias en las dos cadenas de ADN molde de una sola hebra. En estas posiciones de recocido, pueden actuar como cebadores para la Taq ADN polimerasa. Se denominan cebadores porque «preparan» la síntesis de una nueva cadena proporcionando una secuencia corta de ADN de doble cadena para que la Taq polimerasa la extienda y construya una nueva cadena complementaria. La unión de los cebadores a sus secuencias molde también depende en gran medida de la temperatura.
Durante el paso de extensión, el trabajo de la Taq ADN polimerasa es añadir nucleótidos (A, T, G y C) de uno en uno al cebador para crear una copia complementaria de la plantilla de ADN.

Durante la polimerización la temperatura de la reacción es de 72°C, la temperatura que produce la actividad óptima de la Taq polimerasa. Los tres pasos de desnaturalización, extensión y recocido forman un «ciclo» de PCR. Una amplificación de PCR completa se somete a 40 ciclos.
La reacción completa de 40 ciclos se lleva a cabo en un tubo de ensayo colocado en un termociclador. El termociclador contiene un bloque de aluminio que contiene las muestras y puede calentarse y enfriarse rápidamente a través de amplias diferencias de temperatura. El rápido calentamiento y enfriamiento de este bloque térmico se conoce como ciclo de temperatura o ciclo térmico.
Ciclo de temperatura de la PCR = Paso de desnaturalización (94°C) + Paso de recocido (60°C) + Paso de extensión (72°C)
Huellas dactilares de ADN
Número variable de repeticiones en tándem (VNTR)
El principio evolutivo de la variación dentro de una población es una piedra angular en la biología. Esta variación es el resultado de sutiles diferencias en la secuencia de ADN de los individuos de una determinada especie. Uno de los orígenes de la variación se produce cuando se duplica una pequeña secuencia de nucleótidos durante la replicación del ADN. Esto da lugar a una repetición en tándem de la secuencia original. Si este error se produce de nuevo en otra ronda de replicación, entonces tres copias de una secuencia estarán en tándem, como se indica a continuación:
Individuo 1: GTACTCCAATCATGAC
Individuo 2: GTACTCCAATCATCATGAC
Individuo 3: GTACTCCAATCATCATGAC
Aquí se muestran cadenas simples de ADN del mismo locus y de tres individuos diferentes. ‘CAT’ se repite una, dos o tres veces dando lugar a alelos de diferentes longitudes.
Existen dos métodos estándar para la obtención de huellas de ADN:
La PCR permite la amplificación de una sola copia de ADN en millones de copias. Sin embargo, esta técnica requiere que el ADN de interés tenga una secuencia conocida para poder diseñar cebadores que hibriden específicamente con el ADN objetivo. Una de estas regiones que se utiliza en varios países para el análisis forense de muestras de ADN es el locus D1S80. El locus D1S80 está situado en la porción distal del brazo corto del cromosoma 1 y contiene un número variable de repeticiones en tándem.

Ejemplo de repeticiones en tándem D1S80
Utilizando la PCR, se pueden amplificar porciones de ADN que se sabe que contienen VNTRs. El producto resultante se visualiza para obtener el genotipo del ADN de los individuos. Para visualizar el perfil de ADN de un locus determinado, es necesario separar los fragmentos de ADN según su tamaño mediante electroforesis en gel. Se aplica una corriente eléctrica a una matriz de gel de agarosa. Dado que el ADN está cargado negativamente debido a los grupos fosfato, los fragmentos se desplazarán hacia el polo positivo. Las moléculas de ADN de diferentes tamaños migran a través del gel a diferentes velocidades, con los fragmentos más pequeños moviéndose más rápido que los más grandes. Una vez que el ADN se tiñe en el gel, puede visualizarse para revelar el perfil del ADN.

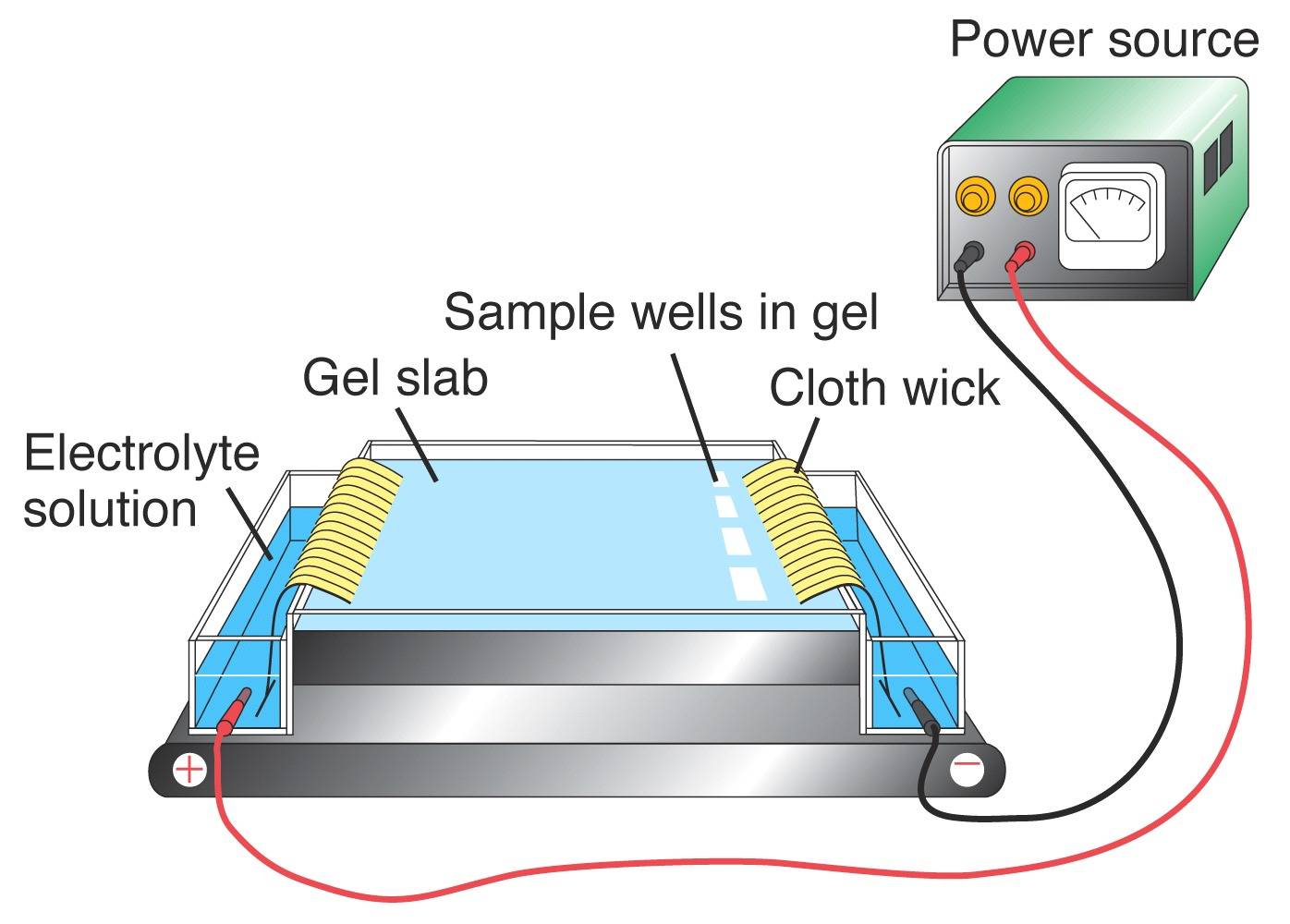
(Izquierda) Gel de agarosa ficticio. Los carriles 1, 2 y 3 representan bandas con los datos de los individuos mencionados anteriormente. El carril 4 representa el ADN de una escena del crimen. (Derecha) Aparato típico de gel de agarosa.
El bromuro de etidio (EtBr) es una molécula básica grande y plana que se asemeja a un par de bases de ADN.

Bromuro de etidio
Debido a su estructura química, puede intercalarse (o insertarse) en una cadena de ADN. El bromuro de etidio se utiliza habitualmente en los laboratorios de biología molecular para teñir geles de electroforesis. El compuesto forma complejos fluorescentes con los ácidos nucleicos y éstos pueden verse bajo la luz ultravioleta. El EtBr se añade a la agarosa calentada antes de verter el gel para obtener una concentración final de 0,5ug/ml. El EtBr es un mutágeno y debe manipularse con extrema precaución utilizando las manos enguantadas.
Secuenciación – Antecedentes
El RFLP (a menudo pronunciado «rif lip», como si fuera una palabra) es un método utilizado por los biólogos moleculares para seguir una secuencia particular de ADN a medida que se transmite a otras células. Los RFLP pueden utilizarse en muchos ámbitos diferentes para lograr distintos objetivos. Los RFLP pueden utilizarse en casos de paternidad o en casos criminales para determinar la fuente de una muestra de ADN. Los RFLP también pueden utilizarse para determinar el estado de la enfermedad de un individuo.
Cada organismo hereda su ADN de sus padres. Dado que el ADN se replica con cada generación, cualquier secuencia dada puede transmitirse a la siguiente generación. Un RFLP es una secuencia de ADN que tiene un sitio de restricción en cada extremo con una secuencia «objetivo» en medio. Una secuencia diana es cualquier segmento de ADN que puede unirse a una sonda formando pares de bases complementarias. Una sonda es una secuencia de ADN monocatenario que ha sido marcada con radiactividad o con una enzima para que la sonda pueda ser detectada. Cuando una sonda se empareja con su objetivo, el investigador puede detectar esta unión y saber dónde está la secuencia objetivo ya que la sonda es detectable. El RFLP produce una serie de bandas cuando se realiza un Southern blot con una combinación particular de enzima de restricción y secuencia de sonda.
Por ejemplo, sigamos un RFLP particular que está definido por la enzima de restricción EcoR I y la secuencia diana de 20 bases GCATGCATGCATGCAT. EcoR I se une a su secuencia de reconocimiento GAATTC y corta el ADN de doble cadena como se muestra:

En el segmento de ADN que se muestra a continuación, se pueden ver los elementos de un RFLP; una secuencia diana flanqueada por un par de sitios de restricción. Cuando este segmento de ADN es cortado por EcoR I, se producen tres fragmentos de restricción, pero sólo uno contiene la secuencia diana que puede ser unida por la secuencia complementaria de la sonda (púrpura).

Veamos dos personas y los segmentos de ADN que llevan que contienen este RFLP (para mayor claridad, sólo veremos uno de los dos puestos de ADN). Como Jack y Jill son organismos diploides (ADN de doble cadena), tienen dos copias de este RFLP. Cuando examinamos una copia de Jack y otra de Jill, vemos que son idénticas:
- Jack 1: -GAATTC—(8,2 kb)—GCATGCATGCAT—(4.2 kb)—GAATTC-
- Jill 1: -GAATTC—(8,2 kb)—GCATGCATGCAT—(4,2 kb)—GAATTC-
- Jack 2: -GAATTC–(1.8 kb)-CCCTTT–(1.2 kb)–GCATGCATGCATGCAT–(1.3 kb)-GAATTC-
- Jill 2: -GAATTC–(1.8 kb)-GAATTC–(1.2 kb)–GCATGCATGCATGCAT–(1.3 kb)-GAATTC-
Cuando examinamos sus segundas copias de este RFLP, vemos que no son idénticas. A Jack 2 le falta un sitio de restricción EcoR I que Jill tiene 1,2 kb aguas arriba de la secuencia objetivo (diferencia en cursiva).
Por tanto, cuando Jack y Jill sometan su ADN al análisis RFLP, tendrán una banda en común y otra que no coincide con la del otro en peso molecular:

Método Southern Blot
Este es un breve resumen de cómo se realiza un Southern blot (más formalmente llamado DNA blot) y qué tipo de datos se pueden obtener de él. El resultado de un Southern Blot es lo que la mayoría de la gente piensa cuando se refiere a una «huella de ADN».

Los Southern blots permiten a los investigadores determinar el peso molecular de un fragmento de restricción y medir las cantidades relativas en diferentes muestras.
Procedimiento:
- El ADN (genómico o de otra fuente) se digiere con una enzima de restricción y se separa mediante electroforesis en gel, normalmente un gel de agarosa. Como hay muchos fragmentos de restricción diferentes en el gel, suele aparecer como una mancha en lugar de bandas discretas. El ADN se desnaturaliza en hebras simples mediante la incubación con NaOH.
- El ADN se transfiere a una membrana que es una hoja de papel secante especial. Los fragmentos de ADN conservan el mismo patrón de separación que tenían en el gel.
- El blot se incuba con muchas copias de una sonda que es ADN monocatenario. Esta sonda formará pares de bases con su secuencia de ADN complementaria y se unirá para formar una molécula de ADN de doble cadena. La sonda no puede verse, pero es radiactiva o tiene una enzima unida a ella (por ejemplo, fosfatasa alcalina o peroxidasa de rábano).
- La localización de la sonda se revela incubándola con un sustrato incoloro que la enzima unida convierte en un producto coloreado que puede verse o que emite luz que expondrá la película de rayos X. Si la sonda estaba marcada con radiactividad, puede exponer la película de rayos X directamente.
A continuación se muestra un ejemplo de un Southern blot real utilizado para detectar la presencia de un gen que se transformó en una población celular mixta. En este Southern blot, es fácil determinar qué células incorporaron el gen y cuáles no.

La figura de la izquierda muestra una fotografía de un gel de agarosa al 0,7% que tiene 14 muestras diferentes cargadas en él (además de un marcador de peso molecular en el carril del extremo derecho y una regla brillante utilizada para analizar los resultados). Cada muestra de ADN ha sido digerida con la misma enzima de restricción (EcoRI). Obsérvese que el ADN no aparece como una serie de bandas discretas, sino como una mancha. La figura de la derecha es una copia de la película de rayos X y revela qué cepas contienen el ADN objetivo y cuáles no.
Un escaneo láser del gel produce una huella genómica del tipo que se muestra a continuación:
