L’empreinte génétique
Comme les empreintes digitales qui ont commencé à être utilisées par les détectives et les laboratoires de police dans les années 1930, chaque personne possède une empreinte génétique unique. Contrairement à l’empreinte digitale classique qui ne se produit que sur le bout des doigts et peut être modifiée par une intervention chirurgicale, l’empreinte génétique est la même pour chaque cellule, tissu et organe d’une personne. Elle ne peut être modifiée par aucun traitement connu. Par conséquent, les empreintes génétiques sont en passe de devenir la principale méthode d’identification et de distinction entre les êtres humains. Les empreintes génétiques sont également utiles dans plusieurs applications de la recherche sur la santé humaine et pour le diagnostic des maladies héréditaires. Les empreintes génétiques sont souvent utilisées dans le système judiciaire pour relier les suspects aux preuves biologiques et résoudre les cas de paternité.
Comment les empreintes génétiques sont-elles utilisées pour identifier un criminel ?
Oxford Education (YouTube)
La réaction en chaîne par polymérase
La PCR a ouvert la voie à une révolution scientifique
En 1983, Kary Mullis, de Cetus Corporation, a mis au point une nouvelle technique, appelée réaction en chaîne par polymérase (PCR). L’objectif de la PCR est de produire une grande quantité d’ADN dans un tube à essai (in vitro), en partant d’une quantité infime. Techniquement parlant, cela signifie la réplication contrôlée d’une séquence d’ADN, ou gène, d’intérêt. Les brins de la matrice peuvent être n’importe quelle forme d’ADN double brin, comme l’ADN génomique. Un chercheur peut prélever des traces d’ADN génomique d’une goutte de sang, d’un follicule pileux ou d’une cellule de la joue (en théorie, un seul brin de matrice suffit pour copier et générer des millions de nouvelles molécules d’ADN identiques) et en produire suffisamment pour les étudier. Avant la PCR, cela aurait été impossible.
Amplification par PCR
On estime qu’il y a 30 000 à 50 000 gènes individuels dans le génome humain. Le véritable pouvoir de la PCR est la capacité de cibler et de faire des millions de copies (ou d’amplifier) d’un morceau d’ADN (ou gène) spécifique à partir d’un génome complet.
La recette d’une amplification d’ADN par PCR contient un mélange simple d’ingrédients qui devraient être reconnaissables comme les morceaux utilisés pour répliquer l’ADN dans le corps :
- Matrice d’ADN – contenant la séquence intacte d’ADN à amplifier
- Désoxynucléotides individuels (A, T, G, et C) – matière première de l’ADN
- L’ADN polymérase – une enzyme qui assemble les nucléotides en une nouvelle chaîne d’ADN
- Les ions magnésium – un cofacteur (catalyseur) nécessaire à l’ADN polymérase pour créer la chaîne d’ADN
- Les amorces oligonucléotidiques. – morceaux d’ADN complémentaires de la matrice qui indiquent à l’ADN polymérase exactement où commencer à faire des copies
- Tampon salé – fournit l’environnement ionique et le pH optimaux pour la réaction PCR
L’ADN matrice peut provenir du sang, de la peau, du sperme ou des cheveux, etc. Lorsque tous les autres composants sont combinés dans les bonnes conditions, une copie de la molécule d’ADN matrice double brin d’origine est fabriquée – ce qui double le nombre de brins de la matrice. Chaque fois que ce cycle est répété, des copies sont faites à partir de copies et le nombre de brins de matrice double – de 2 à 4 à 8 à 16 et ainsi de suite – jusqu’à ce qu’après 20 cycles, il y ait 1 048 554 copies exactes de la séquence cible.
La PCR utilise les mêmes processus de base que les cellules utilisent pour dupliquer leur ADN.
- Hybridation de brins d’ADN complémentaires
- Synthèse de brins d’ADN par l’ADN polymérase
Les amorces d’ADN sont conçues pour flanquer une séquence d’ADN dans le génome et ainsi fournir le signal de départ exact pour que l’ADN polymérase se lie et commence à synthétiser (répliquer) des copies de cet ADN cible. L’hybridation des brins complémentaires a lieu lorsque différentes amorces s’hybrident, ou se lient à chacune de leurs séquences de bases complémentaires respectives sur l’ADN matrice. Les amorces sont de courtes molécules d’ADN monocaténaires (~20 bases de long), l’une étant complémentaire d’une partie du brin 5′-3′, et l’autre étant complémentaire d’une partie du brin 3′-5′ de la matrice. Ces amorces s’hybrident aux brins séparés de la matrice et servent de points de départ à la réplication de l’ADN Taq par l’ADN polymérase.
L’ADN polymérase Taq prolonge les amorces hybridées en » lisant » le brin de la matrice et en synthétisant la séquence complémentaire. De cette façon, la polymérase Taq réplique les deux brins d’ADN matrice. Cette polymérase a été isolée d’une bactérie thermostable (Thermus Aquaticus) qui, dans la nature, vit dans les cheminées de vapeur du parc national de Yellowstone. Pour cette raison, les enzymes au sein de ces bactéries ont évolué pour résister à des températures élevées (94°C) et peuvent être utilisées dans la réaction PCR.
PCR étape par étape
L’amplification par PCR comprend trois étapes principales, une étape de dénaturation, une étape de recuit et une étape d’extension (résumées dans la figure ci-dessous).
1) Dénaturation : Lors de la dénaturation, le mélange réactionnel est chauffé à 94°C pendant 1 minute, ce qui entraîne la fusion ou la séparation de la matrice d’ADN double brin en deux molécules simple brin.
2) Amplification : Dans l’amplification par PCR, les matrices d’ADN doivent être séparées avant que la polymérase puisse générer une nouvelle copie. La température élevée requise pour faire fondre les brins d’ADN devrait normalement détruire l’activité de la plupart des enzymes, mais comme la Taq polymérase a été isolée à partir de bactéries qui se développent dans les températures élevées des sources chaudes, elle reste active.
3) Recuit : Au cours de l’étape de recuit, les amorces oligonucléotidiques « s’annulent » ou trouvent leurs séquences complémentaires sur les deux brins d’ADN de la matrice simple brin. Dans ces positions recuites, elles peuvent agir comme des amorces pour l’ADN polymérase Taq. On les appelle amorces car elles « amorcent » la synthèse d’un nouveau brin en fournissant une courte séquence d’ADN double brin à partir de laquelle la Taq polymérase peut s’étendre et construire un nouveau brin complémentaire. La liaison des amorces à leur séquence matrice est également très dépendante de la température.
Pendant l’étape d’extension, le travail de l’ADN polymérase Taq est d’ajouter des nucléotides (A, T, G et C) un par un à l’amorce pour créer une copie complémentaire de la matrice d’ADN.

Pendant la polymérisation, la température de la réaction est de 72°C, la température qui produit une activité optimale de la Taq polymérase. Les trois étapes de dénaturation, d’extension et de recuit forment un « cycle » de PCR. Une amplification PCR complète subit 40 cycles.
La totalité de la réaction à 40 cycles est réalisée dans un tube à essai placé dans un thermocycleur. Le thermocycleur contient un bloc d’aluminium qui contient les échantillons et peut être rapidement chauffé et refroidi sur de larges différences de température. Le chauffage et le refroidissement rapides de ce bloc thermique sont connus sous le nom de cycle de température ou cycle thermique.
Cycle thermique de PCR = étape de dénaturation (94°C) + étape de recuit (60°C) + étape d’extension (72°C)
Empreinte ADN
Nombre variable de répétitions en tandem (VNTR)
Le principe évolutif de la variation au sein d’une population est une pierre angulaire de la biologie. Cette variation résulte de différences subtiles dans la séquence d’ADN des individus d’une espèce donnée. Une des origines de la variation résulte de la duplication d’une petite séquence de nucléotides lors de la réplication de l’ADN. Il en résulte une répétition en tandem de la séquence originale. Si cette erreur se reproduit lors d’un autre cycle de réplication, alors trois copies d’une séquence seront en tandem, comme indiqué ci-dessous :
Individu 1 : GTACTCCAATCATGTACCATGAC
Individu 2 : GTACTCCAATCATCATGTACCATGAC
Individu 3 : GTACTCCAATCATCATCATGTACCATGAC
Ici sont représentés des brins uniques d’ADN provenant du même locus et de trois individus différents. ‘CAT’ est répété une, deux ou trois fois, ce qui donne des allèles de différentes longueurs.
Il existe deux méthodes standard pour la prise d’empreintes génétiques :
- PCR d’ADN contenant des VNTR.
- Blotting de sud (utilisant des RFLP).
La PCR permet l’amplification d’une seule copie d’ADN en millions de copies. Cependant, cette technique nécessite que l’ADN d’intérêt soit déjà d’une séquence connue afin de concevoir des amorces qui s’hybrideront spécifiquement à l’ADN cible. L’une de ces régions, utilisée dans plusieurs pays pour l’analyse médico-légale d’échantillons d’ADN, est le locus D1S80. Le locus D1S80 est situé sur la partie distale du bras court du chromosome 1 et contient un nombre variable de répétitions en tandem.

D1S80 Exemple de répétitions en tandem
Electrophorèse
En utilisant la PCR, des portions d’ADN connues pour contenir des VNTR peuvent être amplifiées. Le produit résultant est ensuite visualisé pour obtenir le génotype ADN des individus. Afin de visualiser le profil d’ADN d’un locus donné, les fragments d’ADN doivent être séparés en fonction de leur taille par électrophorèse sur gel. Un courant électrique est appliqué à une matrice de gel d’agarose. L’ADN étant chargé négativement en raison des groupes phosphates, les fragments se déplacent vers le pôle positif. Les molécules d’ADN de différentes tailles migrent à travers le gel à des vitesses différentes, les fragments les plus petits se déplaçant plus rapidement que les plus grands. Une fois l’ADN coloré dans le gel, il peut être visualisé pour révéler le profil de l’ADN.

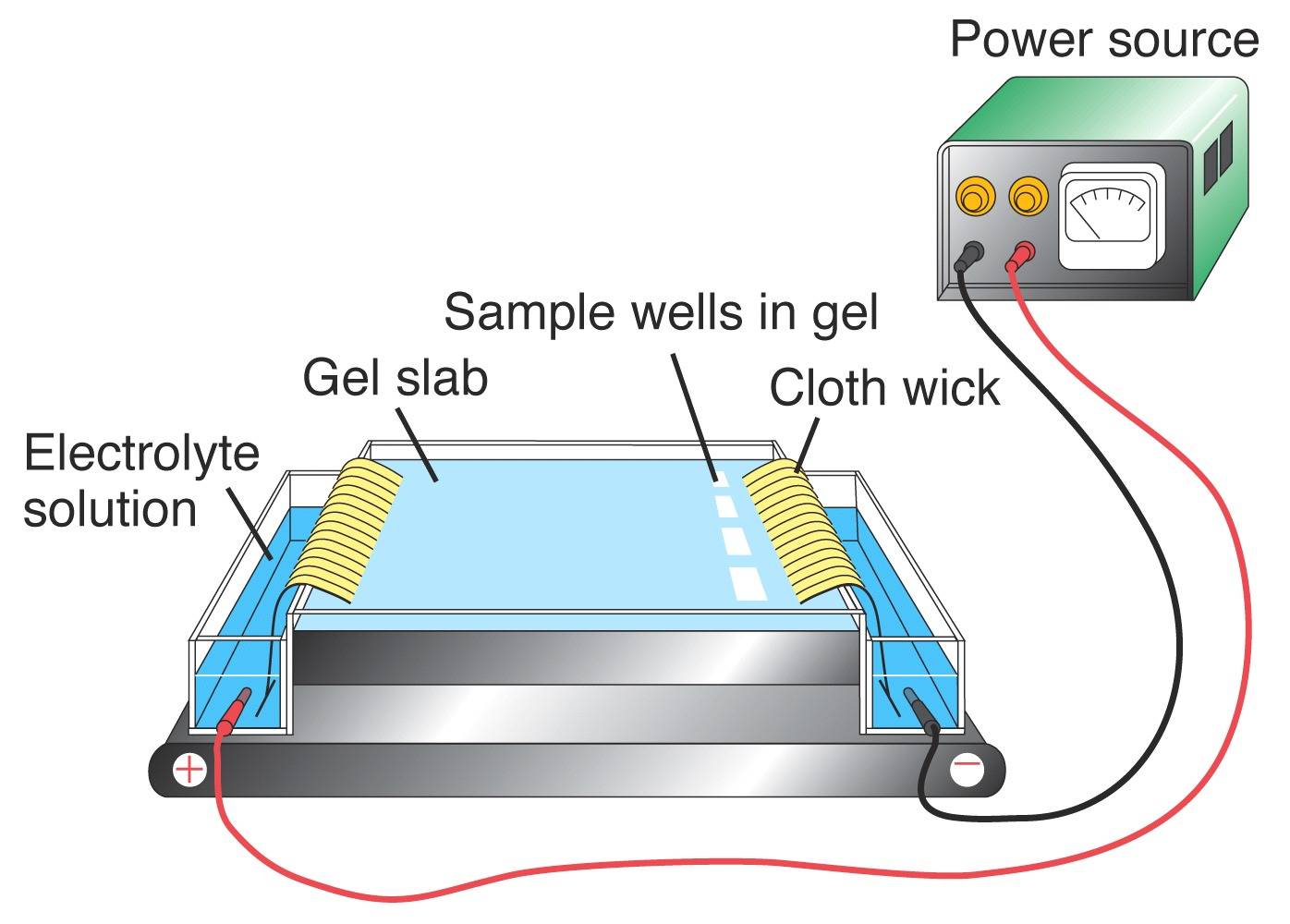
(A gauche) Gel d’agarose fictif. Les voies 1, 2 et 3 représentent des bandes utilisant les données des individus cités ci-dessus. Le couloir 4 représente l’ADN d’une scène de crime. (Droite) Appareil typique de gel d’agarose.
Le bromure d’éthidium (EtBr) est une grande molécule basique plate qui ressemble à une paire de bases d’ADN.

Bromure d’éthidium
En raison de sa structure chimique, il peut s’intercaler (ou s’insérer) dans un brin d’ADN. Le bromure d’éthidium est couramment utilisé dans les laboratoires de biologie moléculaire pour colorer les gels d’électrophorèse. Ce composé forme des complexes fluorescents avec les acides nucléiques et ceux-ci peuvent être visualisés sous la lumière UV. L’EtBr est ajouté à l’agarose chauffé avant de verser le gel pour obtenir une concentration finale de 0,5ug/ml. L’EtBr est un mutagène et doit être manipulé avec une extrême prudence en utilisant des mains gantées.
Séquençage – Contexte
La RFLP (souvent prononcée « rif lip », comme si c’était un mot) est une méthode utilisée par les biologistes moléculaires pour suivre une séquence particulière d’ADN lorsqu’elle est transmise à d’autres cellules. Les RFLP peuvent être utilisées dans de nombreux contextes différents pour atteindre différents objectifs. Les RFLP peuvent être utilisées dans des affaires de paternité ou des affaires criminelles pour déterminer la source d’un échantillon d’ADN. Les RFLP peuvent également être utilisées pour déterminer le statut de la maladie d’un individu.
Chaque organisme hérite de son ADN de ses parents. L’ADN étant répliqué à chaque génération, toute séquence donnée peut être transmise à la génération suivante. Un RFLP est une séquence d’ADN qui comporte un site de restriction à chaque extrémité, avec une séquence « cible » entre les deux. Une séquence cible est tout segment d’ADN qui peut se lier à une sonde en formant des paires de bases complémentaires. Une sonde est une séquence d’ADN simple brin qui a été marquée avec de la radioactivité ou une enzyme afin que la sonde puisse être détectée. Lorsqu’une sonde se lie par paires de bases à sa cible, l’enquêteur peut détecter cette liaison et savoir où se trouve la séquence cible puisque la sonde est détectable. La RFLP produit une série de bandes lorsqu’un transfert de Southern est effectué avec une combinaison particulière d’enzyme de restriction et de séquence de sonde.
Par exemple, suivons une RFLP particulière qui est définie par l’enzyme de restriction EcoR I et la séquence cible de 20 bases GCATGCATGCATGCATGCAT. EcoR I se lie à sa séquence de reconnaissance GAATTC et coupe l’ADN double brin comme indiqué :

Dans le segment d’ADN présenté ci-dessous, vous pouvez voir les éléments d’une RFLP ; une séquence cible flanquée d’une paire de sites de restriction. Lorsque ce segment d’ADN est coupé par EcoR I, trois fragments de restriction sont produits, mais un seul contient la séquence cible qui peut être liée par la séquence sonde complémentaire (en violet).

Regardons deux personnes et les segments d’ADN qu’elles portent et qui contiennent ce RFLP (pour plus de clarté, nous ne verrons qu’un seul des deux peuplements d’ADN). Comme Jack et Jill sont tous deux des organismes diploïdes (ADN double brin), ils possèdent deux copies de ce RFLP. Lorsque nous examinons une copie de Jack et une copie de Jill, nous voyons qu’elles sont identiques :
- Jack 1 : -GAATTC—(8,2 kb)—GCATGCATGCATGCATGCAT—(4.2 kb)—GAATTC-
- Jill 1 : -GAATTC—(8,2 kb)—GCATGCATGCATGCAT—(4,2 kb)—GAATTC-
Lorsque nous examinons leurs secondes copies de cette RFLP, nous voyons qu’elles ne sont pas identiques. Il manque à Jack 2 un site de restriction EcoR I que Jill possède 1,2 kb en amont de la séquence cible (différence en italique).
- Jack 2 : -GAATTC–(1.8 kb)-CCCTTT–(1.2 kb)–GCATGCATGCATGCATGCAT–(1.3 kb)-GAATTC-
- Jill 2 : -GAATTC–(1.8 kb)-GAATTC–(1.2 kb)–GCATGCATGCATGCAT–(1.3 kb)-GAATTC-
Par conséquent, lorsque Jack et Jill verront leur ADN soumis à une analyse RFLP, ils auront une bande en commun et une bande qui ne correspond pas à celle de l’autre en termes de poids moléculaire :

Méthode du Southern Blot
Ceci est un bref aperçu de la façon dont un Southern Blot (plus formellement appelé DNA Blot) est réalisé et du type de données que vous pouvez obtenir à partir de celui-ci. Le résultat d’un Southern Blot est ce à quoi la plupart des gens pensent lorsqu’ils font référence à une « empreinte génétique ».

Les Southern blots permettent aux enquêteurs de déterminer le poids moléculaire d’un fragment de restriction et de mesurer les quantités relatives dans différents échantillons.
Procédure:
- L’ADN (génomique ou autre source) est digéré avec une enzyme de restriction et séparé par électrophorèse sur gel, généralement un gel d’agarose. Comme il y a beaucoup de fragments de restriction différents sur le gel, il apparaît généralement comme un frottis plutôt que des bandes discrètes. L’ADN est dénaturé en brins uniques par incubation avec du NaOH.
- L’ADN est transféré sur une membrane qui est une feuille de papier buvard spécial. Les fragments d’ADN conservent le même schéma de séparation qu’ils avaient sur le gel.
- Le buvard est incubé avec de nombreuses copies d’une sonde qui est de l’ADN monocaténaire. Cette sonde va former des paires de bases avec sa séquence d’ADN complémentaire et se lier pour former une molécule d’ADN double brin. La sonde ne peut pas être vue mais elle est soit radioactive, soit liée à une enzyme (par exemple la phosphatase alcaline ou la peroxydase de raifort).
- L’emplacement de la sonde est révélé en l’incubant avec un substrat incolore que l’enzyme fixée convertit en un produit coloré que l’on peut voir ou qui émet de la lumière qui exposera le film radiographique. Si la sonde a été marquée avec de la radioactivité, elle peut exposer directement un film à rayons X.
Vous trouverez ci-dessous un exemple de véritable transfert de Southern utilisé pour détecter la présence d’un gène qui a été transformé dans une population cellulaire mixte. Dans ce Southern blot, il est facile de déterminer quelles cellules ont incorporé le gène et celles qui ne l’ont pas fait.

La figure de gauche montre une photographie d’un gel d’agarose à 0,7 % sur lequel sont chargés 14 échantillons différents (plus un marqueur de poids moléculaire dans la voie d’extrême droite et une règle lumineuse utilisée pour l’analyse des résultats). Chaque échantillon d’ADN a été digéré avec la même enzyme de restriction (EcoRI). Remarquez que l’ADN n’apparaît pas sous la forme d’une série de bandes discrètes mais plutôt d’un frottis. La figure de droite est une copie du film radiographique et révèle les souches qui contiennent l’ADN cible et celles qui ne le contiennent pas.
Un balayage laser du gel produit une empreinte génomique du type ci-dessous :

.