L’impronta digitale del DNA
Come le impronte digitali che entrarono in uso dai detective e dai laboratori di polizia negli anni ’30, ogni persona ha un’impronta digitale unica del DNA. A differenza di un’impronta digitale convenzionale che si trova solo sulla punta delle dita e può essere alterata da un intervento chirurgico, un’impronta del DNA è la stessa per ogni cellula, tessuto e organo di una persona. Non può essere alterata da nessun trattamento conosciuto. Di conseguenza, l’impronta del DNA sta diventando rapidamente il metodo principale per identificare e distinguere tra i singoli esseri umani. Le impronte digitali del DNA sono anche utili in diverse applicazioni della ricerca sanitaria umana e per la diagnosi di disturbi ereditari. L’impronta del DNA è spesso usata nel sistema giudiziario per collegare i sospetti alle prove biologiche e risolvere i casi di paternità.
Come viene usata l’impronta del DNA per identificare un criminale?
Oxford Education (YouTube)
La reazione a catena della polimerasi
PCR ha posto le basi per una rivoluzione scientifica
Nel 1983, Kary Mullis della Cetus Corporation sviluppò una nuova tecnica, chiamata reazione a catena della polimerasi (PCR). L’obiettivo della PCR è quello di produrre una grande quantità di DNA in una provetta (in vitro), partendo da una quantità minima. Tecnicamente parlando, questo significa la replicazione controllata di una sequenza di DNA, o gene, di interesse. I filamenti modello possono essere qualsiasi forma di DNA a doppio filamento come il DNA genomico. Un ricercatore può prendere tracce di DNA genomico da una goccia di sangue, da un singolo follicolo pilifero o da una cellula della guancia (in teoria, è necessario un solo filamento modello per copiare e generare milioni di nuove molecole di DNA identiche) e farne abbastanza per studiare. Prima della PCR, questo sarebbe stato impossibile.
Amplificazione PCR
Si stima che ci siano 30.000-50.000 geni individuali nel genoma umano. Il vero potere della PCR è la capacità di mirare e fare milioni di copie di (o amplificare) un pezzo specifico di DNA (o gene) da un genoma completo.
La ricetta per un’amplificazione PCR del DNA contiene una semplice miscela di ingredienti che dovrebbero essere riconoscibili come i pezzi usati per replicare il DNA nel corpo:
- Modello di DNA – contenente la sequenza intatta di DNA da amplificare
- Deossinucleotidi individuali (A, T, G, e C) – materia prima del DNA
- DNA polimerasi – un enzima che assembla i nucleotidi in una nuova catena di DNA
- Ioni magnesio – un cofattore (catalizzatore) richiesto dalla DNA polimerasi per creare la catena di DNA
- Imprimitori oligonucleotidici – pezzi di DNA complementari al modello che dicono alla DNA polimerasi esattamente dove iniziare a fare copie
- Buffer di sale – fornisce l’ambiente ionico ottimale e il pH per la reazione PCR
Il DNA modello può essere dal sangue, pelle, sperma o capelli, ecc. Quando tutti gli altri componenti sono combinati nelle giuste condizioni, viene fatta una copia della molecola di DNA modello originale a doppio filamento – raddoppiando il numero di filamenti modello. Ogni volta che questo ciclo viene ripetuto, le copie sono fatte da copie e il numero di filamenti modello raddoppia – da 2 a 4 a 8 a 16 e così via – finché dopo 20 cicli ci sono 1.048.554 copie esatte della sequenza target.
PCR fa uso degli stessi processi di base che le cellule usano per duplicare il loro DNA.
- Ibridizzazione del filamento di DNA complementare
- Sintesi del filamento di DNA tramite la DNA polimerasi
I primer del DNA sono progettati per fiancheggiare una sequenza di DNA all’interno del genoma e quindi fornire l’esatto segnale di inizio per la DNA polimerasi per legarsi e iniziare a sintetizzare (replicare) copie di quel DNA target. L’ibridazione del filamento complementare avviene quando diversi primer si annebbiano, o si legano a ciascuna delle loro rispettive sequenze di basi complementari sul DNA modello. I primer sono brevi molecole di DNA a singolo filamento (lunghe ~ 20 basi), uno complementare a una porzione del filamento 5′-3′ e un altro complementare a una porzione del filamento 3′-5′ del modello. Questi inneschi si riconnettono ai filamenti separati del modello e servono come punti di partenza per la replicazione del DNA Taq da parte della DNA polimerasi.
La DNA polimerasi Taq estende gli inneschi riconnessi “leggendo” il filamento del modello e sintetizzando la sequenza complementare. In questo modo, la Taq polimerasi replica i due filamenti di DNA modello. Questa polimerasi è stata isolata da un batterio termostabile (Thermus Aquaticus) che in natura vive all’interno delle bocche di vapore del Parco Nazionale di Yellowstone. Per questo motivo gli enzimi all’interno di questi batteri si sono evoluti per resistere alle alte temperature (94°C) e possono essere usati nella reazione PCR.
PCR Step by Step
L’amplificazione PCR include tre fasi principali, una fase di denaturazione, una fase di ricottura e una fase di estensione (riassunta nella figura sottostante).
1) Denaturazione: Nella denaturazione la miscela di reazione viene riscaldata a 94°C per 1 minuto, il che provoca la fusione o la separazione del modello di DNA a doppio filamento in due molecole a filamento singolo.
2) Amplificazione: Nell’amplificazione PCR, i modelli di DNA devono essere separati prima che la polimerasi possa generare una nuova copia. L’alta temperatura richiesta per fondere i filamenti di DNA normalmente distruggerebbe l’attività della maggior parte degli enzimi, ma poiché la Taq polimerasi è stata isolata da batteri che prosperano nelle alte temperature delle sorgenti calde, rimane attiva.
3) Ricottura: Durante la fase di annealing, i primer oligonucleotidici “anneal to” o trovano le loro sequenze complementari sui due filamenti template a singolo filamento di DNA. In queste posizioni di annealing, possono agire come primer per la Taq DNA polimerasi. Sono chiamati primer perché “innescano” la sintesi di un nuovo filamento fornendo una breve sequenza di DNA a doppio filamento da cui la Taq polimerasi può estendere e costruire un nuovo filamento complementare. Il legame dei primer alle loro sequenze di template è anche altamente dipendente dalla temperatura.
Durante la fase di estensione, il compito della Taq DNA polimerasi è quello di aggiungere nucleotidi (A, T, G e C) uno alla volta al primer per creare una copia complementare del modello di DNA.

Durante la polimerizzazione la temperatura di reazione è 72°C, la temperatura che produce un’attività ottimale della Taq polimerasi. Le tre fasi di denaturazione, estensione e ricottura formano un “ciclo” di PCR. Un’amplificazione PCR completa è sottoposta a 40 cicli.
L’intera reazione di 40 cicli viene effettuata in una provetta posta in un termociclatore. Il termociclatore contiene un blocco di alluminio che contiene i campioni e può essere rapidamente riscaldato e raffreddato attraverso ampie differenze di temperatura. Il rapido riscaldamento e raffreddamento di questo blocco termico è noto come ciclo di temperatura o ciclo termico.
PCR Temperature Cycle = Step di Denaturazione (94°C) + Step di Ricottura (60°C) + Step di Estensione (72°C)
DNA Fingerprinting
Variable Number of Tandem Repeats (VNTR)
Il principio evolutivo della variazione all’interno di una popolazione è una pietra miliare della biologia. Questa variazione deriva da sottili differenze nella sequenza del DNA negli individui di una data specie. Un’origine di variazione risulta quando la duplicazione di una piccola sequenza di nucleotidi avviene durante la replicazione del DNA. Questo risulta in una ripetizione in tandem della sequenza originale. Se questo errore si verifica di nuovo in un altro giro di replicazione, allora tre copie di una sequenza saranno in tandem, come indicato di seguito:
Individuo 1: GTACTCCAATCATGTACCATGAC
Individuo 2: GTACTCCAATCATCATGTACCATGAC
Individuo 3: GTACTCCAATCATCATCATGTACCATGAC
Qui sono mostrati singoli filamenti di DNA dallo stesso locus e tre individui diversi. CAT’ è ripetuto una, due o tre volte dando luogo ad alleli di diversa lunghezza.
Ci sono due metodi standard per il DNA fingerprinting:
- PCR del DNA contenente VNTRs.
- Southern blotting (usando RFLPs).
PCR permette l’amplificazione di una singola copia di DNA in milioni di copie. Tuttavia questa tecnica richiede che il DNA di interesse sia già di una sequenza nota per progettare primer che si ibridizzino specificamente con il DNA target. Una di queste regioni, utilizzata in diversi paesi per l’analisi forense di campioni di DNA, è il locus D1S80. Il locus D1S80 si trova sulla porzione distale del braccio corto del cromosoma 1 e contiene un numero variabile di ripetizioni in tandem.

D1S80 Esempio di ripetizioni in tandem
Elettroforesi
Utilizzando la PCR, porzioni di DNA che sono note per contenere VNTR possono essere amplificate. Il prodotto risultante viene poi visualizzato per ottenere il genotipo del DNA dell’individuo. Per visualizzare il profilo del DNA di un dato locus, i frammenti di DNA devono essere separati in base alle loro dimensioni utilizzando l’elettroforesi su gel. Una corrente elettrica viene applicata ad una matrice di gel di agarosio. Poiché il DNA è caricato negativamente a causa dei gruppi fosfato, i frammenti si muoveranno verso il polo positivo. Le molecole di DNA di dimensioni diverse migrano attraverso il gel a velocità diverse, con i frammenti più piccoli che si muovono più velocemente di quelli più grandi. Una volta che il DNA è colorato nel gel, può essere visualizzato per rivelare il profilo del DNA.

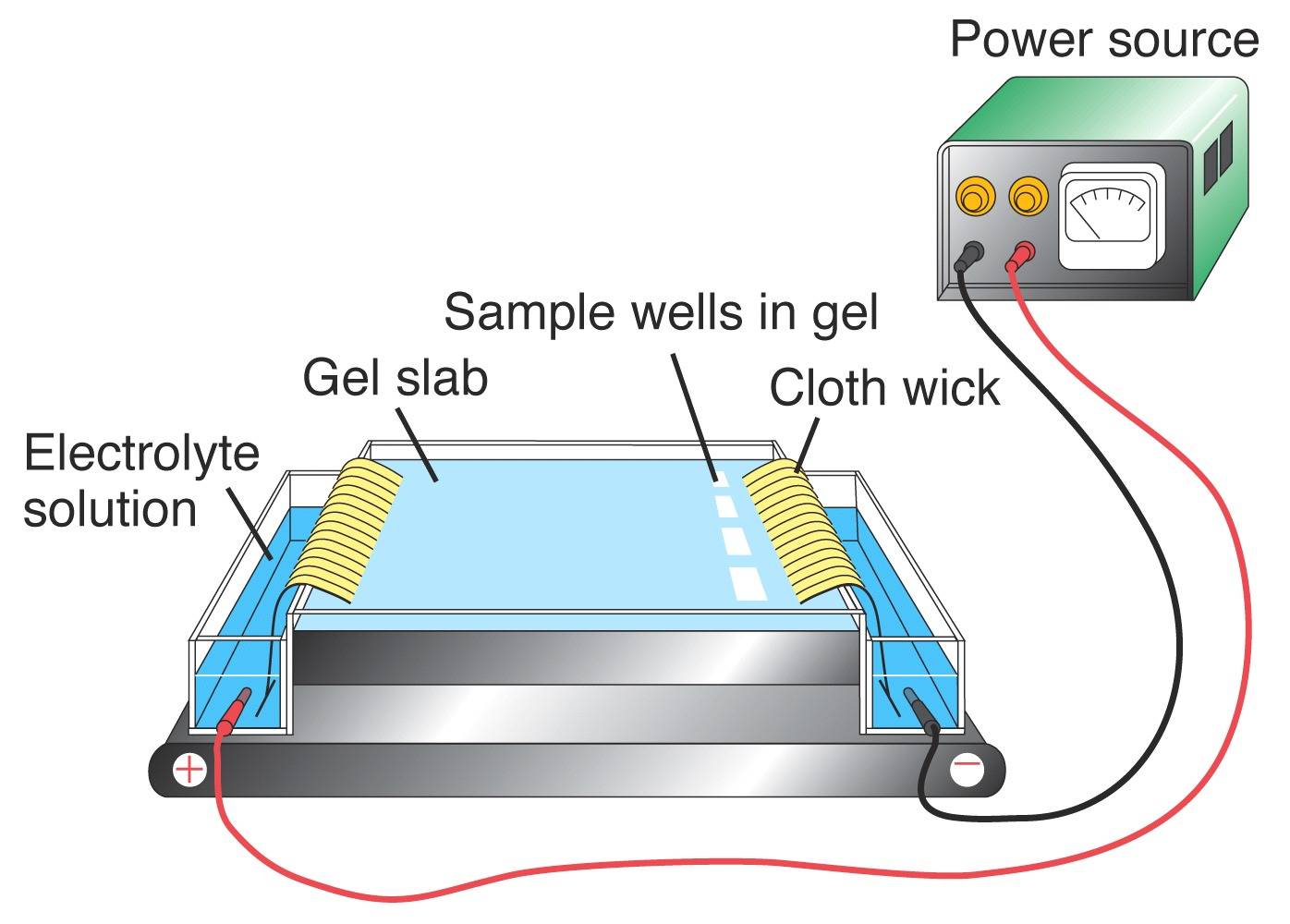
(A sinistra) Gel di agarosio fittizio. Le corsie 1, 2 e 3 rappresentano le bande utilizzando i dati degli individui elencati sopra. La corsia 4 rappresenta il DNA di una scena del crimine. (Destra) Apparato tipico del gel di agarosio.
Il bromuro di etidio (EtBr) è una molecola di base grande e piatta che assomiglia a una coppia di basi del DNA.

Bromuro di Etidio
A causa della sua struttura chimica, può intercalarsi (o inserirsi) in un filamento di DNA. Il bromuro di etidio è comunemente usato nei laboratori di biologia molecolare per colorare i gel di elettroforesi. Il composto forma complessi fluorescenti con gli acidi nucleici e questi possono essere visti sotto la luce UV. L’EtBr viene aggiunto all’agarosio riscaldato prima di versare il gel per dare una concentrazione finale di 0.5ug/ml. L’EtBr è un mutageno e deve essere maneggiato con estrema cautela usando mani guantate.
Sequencing – Background
RFLP (spesso pronunciato “rif lip”, come se fosse una parola) è un metodo usato dai biologi molecolari per seguire una particolare sequenza di DNA mentre viene passata ad altre cellule. Le RFLP possono essere usate in molti contesti diversi per raggiungere diversi obiettivi. Gli RFLP possono essere usati nei casi di paternità o nei casi criminali per determinare la fonte di un campione di DNA. Le RFLP possono anche essere usate per determinare lo stato di malattia di un individuo.
Ogni organismo eredita il suo DNA dai suoi genitori. Poiché il DNA si replica ad ogni generazione, qualsiasi sequenza può essere trasmessa alla generazione successiva. Un RFLP è una sequenza di DNA che ha un sito di restrizione ad ogni estremità con una sequenza “target” in mezzo. Una sequenza bersaglio è qualsiasi segmento di DNA che può legarsi a una sonda formando coppie di basi complementari. Una sonda è una sequenza di DNA a filamento singolo che è stata marcata con radioattività o con un enzima in modo che la sonda possa essere rilevata. Quando una sonda si accoppia con il suo bersaglio, lo sperimentatore può rilevare questo legame e sapere dove si trova la sequenza bersaglio poiché la sonda è rilevabile. La RFLP produce una serie di bande quando si esegue un Southern blot con una particolare combinazione di enzima di restrizione e sequenza di sonda.
Per esempio, seguiamo una particolare RFLP che è definita dall’enzima di restrizione EcoR I e la sequenza target di 20 basi GCATGCATGCATGCATGCAT. EcoR I si lega alla sua sequenza di riconoscimento GAATTC e taglia il DNA a doppio filamento come mostrato:

Nel segmento di DNA mostrato sotto, si possono vedere gli elementi di una RFLP; una sequenza bersaglio affiancata da una coppia di siti di restrizione. Quando questo segmento di DNA viene tagliato da EcoR I, vengono prodotti tre frammenti di restrizione, ma solo uno contiene la sequenza bersaglio che può essere legata dalla sequenza sonda complementare (viola).

Guardiamo due persone e i segmenti di DNA che portano che contengono questa RFLP (per chiarezza, vedremo solo uno dei due campioni di DNA). Poiché Jack e Jill sono entrambi organismi diploidi (DNA a doppio filamento), hanno due copie di questo RFLP. Quando esaminiamo una copia di Jack e una copia di Jill, vediamo che sono identiche:
- Jack 1: -GAATTC—(8,2 kb)—GCATGCATGATGCATGATGCAT—(4.2 kb)—GAATTC-
- Jill 1: -GAATTC—(8.2 kb)—GCATGCATGCATGCATGCAT—(4.2 kb)—GAATTC-
Quando esaminiamo le loro seconde copie di questo RFLP, vediamo che non sono identiche. A Jack 2 manca un sito di restrizione EcoR I che Jill ha 1,2 kb a monte della sequenza target (differenza in corsivo).
- Jack 2: -GAATTC–(1.8 kb)-CCCTTT–(1.2 kb)–GCATGCATGCATGCATGCATGCAT–(1.3 kb)-GAATTC-
- Jill 2: -GAATTC–(1.8 kb)-GAATTC–(1.2 kb)–GCATGCATGCATGCAT–(1.3 kb)-GAATTC-
Quindi, quando Jack e Jill sottopongono il loro DNA all’analisi RFLP, avranno una banda in comune e una banda che non corrisponde a quella dell’altro in peso molecolare:

Metodo Southern Blot
Questa è una breve panoramica di come viene eseguito un Southern blot (più formalmente chiamato DNA blot) e che tipo di dati si possono ottenere. Il risultato di un Southern Blot è quello a cui la maggior parte delle persone pensa quando si riferisce ad una “impronta digitale del DNA”.

Il Southern blot permette ai ricercatori di determinare il peso molecolare di un frammento di restrizione e di misurare le quantità relative in diversi campioni.
Procedura:
- Il DNA (genomico o di altra fonte) viene digerito con un enzima di restrizione e separato mediante elettroforesi su gel, solitamente un gel di agarosio. Poiché ci sono molti frammenti di restrizione diversi sul gel, di solito appare come uno striscio piuttosto che bande discrete. Il DNA viene denaturato in singoli filamenti mediante incubazione con NaOH.
- Il DNA viene trasferito su una membrana che è un foglio di carta speciale. I frammenti di DNA mantengono lo stesso schema di separazione che avevano sul gel.
- Il blot viene incubato con molte copie di una sonda che è DNA a singolo filamento. Questa sonda formerà coppie di basi con la sua sequenza di DNA complementare e si legherà per formare una molecola di DNA a doppio filamento. La sonda non può essere vista, ma è radioattiva o ha un enzima legato ad essa (ad esempio fosfatasi alcalina o perossidasi di rafano).
- La posizione della sonda viene rivelata incubandola con un substrato incolore che l’enzima collegato converte in un prodotto colorato che può essere visto o emette luce che esporrà la pellicola a raggi X. Se la sonda è stata etichettata con radioattività, può esporre direttamente la pellicola a raggi X.
Di seguito è riportato un esempio di un vero Southern blot utilizzato per rilevare la presenza di un gene che è stato trasformato in una popolazione di cellule miste. In questo Southern blot, è facile determinare quali cellule hanno incorporato il gene e quali no.

La figura a sinistra mostra una fotografia di un gel di agarosio allo 0,7% che ha 14 diversi campioni caricati su di esso (più il marker del peso molecolare nella corsia all’estrema destra e un righello luminoso usato per l’analisi dei risultati). Ogni campione di DNA è stato digerito con lo stesso enzima di restrizione (EcoRI). Notate che il DNA non appare come una serie di bande discrete ma piuttosto come uno striscio. La figura a destra è una copia della pellicola a raggi X e rivela quali ceppi contengono il DNA bersaglio e quali no.
Una scansione laser del gel produce un’impronta genomica del tipo seguente:
