DNA指紋
1930年代に探偵や警察が使用し始めた指紋のように、人はそれぞれ固有のDNA指紋を持っています。 従来の指紋は、指先にのみ存在し、手術によって変えることができましたが、DNA指紋は、人のすべての細胞、組織、器官で同じです。 どんな治療法でも変更することはできません。 そのため、DNA指紋は、人間を識別するための主要な方法として急速に普及しています。 また、DNA指紋は、人間の健康管理に関する研究や、遺伝性疾患の診断にも役立ちます。
How is DNA fingerprinting used to identification a criminal?
Oxford Education (YouTube)
The Polymerase Chain Reaction
PCR Set the Stage for a Scientific Revolution
1983年、Cetus CorporationのKary Mullis氏は、Polymerase Chain Reaction (PCR)と呼ばれる新しい技術を開発しました。 PCRの目的は、試験管の中で(in vitro)、わずかな量から大量のDNAを作り出すことです。 技術的には、目的のDNA配列または遺伝子の複製を制御することを意味します。 鋳型となるのは、ゲノムDNAのような二本鎖のDNAであれば何でもいい。 研究者は、1滴の血液、1つの毛包、または頬の細胞から微量のゲノムDNAを採取し(理論的には、数百万の新しい同一のDNA分子をコピーして生成するためには、たった1本のテンプレート鎖が必要です)、研究に必要な量を作ることができます。
PCRによる増幅
ヒトのゲノムには、3万〜5万個の遺伝子があると言われています。 PCRの真の力は、完全なゲノムから特定のDNA(または遺伝子)を狙って何百万コピーも作る(または増幅する)能力です。
DNAのPCR増幅のレシピには、体内でDNAを複製するのに使用されるピースとして認識できるような、シンプルな成分の混合物が含まれています。
- DNAテンプレート – 増幅されるべきDNAのそのままの配列を含む
- 個々のデオキシヌクレオチド(A、T、G。 DNAの原料
- DNAポリメラーゼ – ヌクレオチドを新しいDNA鎖に組み立てる酵素
- マグネシウムイオン – DNAポリメラーゼがDNA鎖を作るのに必要な補因子(触媒)
- オリゴヌクレオチドプライマー – 鋳型に相補的なDNAの断片で、DNAポリメラーゼにどこからコピーを始めるかを正確に伝えるもの
- 塩緩衝液 – PCR反応に最適なイオン環境とpHを提供するもの
鋳型となるDNAは、血液、皮膚、精液、毛髪などから採取することができます。 鋳型となるDNAは、血液、皮膚、精液、髪の毛などから採取します。 他のすべての成分が適切な条件で組み合わされると、元の二本鎖の鋳型DNA分子のコピーが作られ、鋳型の鎖の数が2倍になります。
PCRは、細胞がDNAを複製するのと同じ基本的なプロセスを利用しています。
- 相補的なDNA鎖のハイブリダイゼーション
- DNAポリメラーゼによるDNA鎖の合成
DNAプライマーは、ゲノム内のDNA配列を挟むように設計されているため、DNAポリメラーゼが結合し、標的DNAのコピーの合成(複製)を開始するための正確なスタートシグナルとなります。 相補鎖ハイブリダイゼーションは、異なるプライマーがテンプレートDNA上のそれぞれの相補的な塩基配列にアニーリング(結合)することで起こる。 プライマーは短い一本鎖のDNA分子(長さ約20塩基)で、1つは5′-3’鎖の一部に相補的であり、もう1つは鋳型の3′-5’鎖の一部に相補的である。
Taq DNAポリメラーゼは、鋳型の鎖を「読み」、相補的な配列を合成することで、アニールされたプライマーを伸長する。 このようにして、Taqポリメラーゼは2本のテンプレートDNA鎖を複製します。 このポリメラーゼは、自然界ではイエローストーン国立公園の蒸気噴出孔に生息する熱安定性細菌(Thermus Aquaticus)から分離されたものである。
PCR Step by Step
PCRの増幅には、大きく分けて変性、アニーリング、伸長の3つのステップがあります(下図参照)。
1) 変性。 変性では、反応混合物を94℃で1分間加熱することで、二本鎖のDNAテンプレートが融解または分離し、2本の一本鎖分子になります。
2) 増幅。 PCRによる増幅では、ポリメラーゼが新しいコピーを生成する前に、DNAテンプレートを分離する必要があります。
3)アニーリング:DNAを溶かすのに必要な高温によって、ほとんどの酵素は活性を失ってしまうが、Taqポリメラーゼは高温の温泉で繁殖するバクテリアから分離されたため、活性を維持することができる。 アニーリング工程では、オリゴヌクレオチドプライマーは、2本の一本鎖の鋳型DNA上に相補的な配列を見つけます。 このアニーリングした位置で、Taq DNAポリメラーゼのプライマーとして働くことができる。 プライマーと呼ばれるのは、Taqポリメラーゼが伸長して新しい相補的な鎖を作るために、二本鎖DNAの短い配列を提供することで、新しい鎖の合成を「プライム」するからである。 プライマーの鋳型配列への結合は、温度にも大きく依存します。
伸長ステップでは、Taq DNAポリメラーゼの仕事は、プライマーにヌクレオチド(A、T、G、C)を一度に1つずつ加えて、DNAテンプレートの相補的コピーを作ることです。

重合中の反応温度は72℃で、最適なTaqポリメラーゼの活性が得られる温度です。 変性、伸長、アニーリングの3つのステップでPCRの1つの「サイクル」を形成します。
40サイクルの反応は、サーマルサイクラーに入れた試験管の中で行われます。 サーマルサイクラーには、サンプルを入れるアルミブロックがあり、広い温度差で急速に加熱・冷却することができます。 このサーマルブロックの急速な加熱・冷却は、温度サイクリングまたはサーマルサイクリングと呼ばれています。
PCR Temperature Cycle = Denaturation Step (94°C) + Annealing Step (60°C) + Extension Step (72°C)
div
DNAフィンガープリンティング
可変数タンデムリピート(VNTR)
集団内での変異という進化の原理は、生物学の基礎となるものです。 この変動は、ある種の個体におけるDNA配列の微妙な違いに起因します。 DNAの複製の際に、小さな塩基配列が重複してしまうことが、変異の原因の一つとされています。 その結果、元の配列がタンデムに繰り返されることになります。
個体1:GTACTCCAATCATGTACCATGAC
個体2:GTACTCCAATCATGTACCATGAC
個体3:GTACTCCAATCATCATGTACCATGAC
ここでは、同じ遺伝子座と3つの異なる個体から採取したDNAの一本鎖を示しています。
DNAフィンガープリントには2つの標準的な方法があります:
- VNTRを含むDNAのPCR
- サザンブロッティング(RFLPを使用)
。
PCRは、1コピーのDNAを数百万コピーに増幅することができます。 しかし、この技術では、対象となるDNAに特異的にハイブリダイズするプライマーを設計するために、対象となるDNAがすでに既知の配列である必要があります。 いくつかの国でDNAサンプルの法医学的分析に使用されているそのような領域の一つがD1S80遺伝子座である。

D1S80 Example Tandem Repeats
Electrophoresis
PCRを使って、VNTRを含むことがわかっているDNAの一部を増幅することができます。 その結果、得られた産物を見ることで、個人のDNA遺伝子型を得ることができます。 また、ある遺伝子座のDNAプロファイルを可視化するためには、ゲル電気泳動でDNA断片をサイズごとに分離する必要があります。 アガロースゲルのマトリックスに電流を流します。 DNAはリン酸基の影響で負の電荷を帯びているため、断片は正の極に向かって移動する。 異なるサイズのDNA分子は、異なる速度でゲル内を移動し、小さな断片は大きな断片よりも速く移動します。

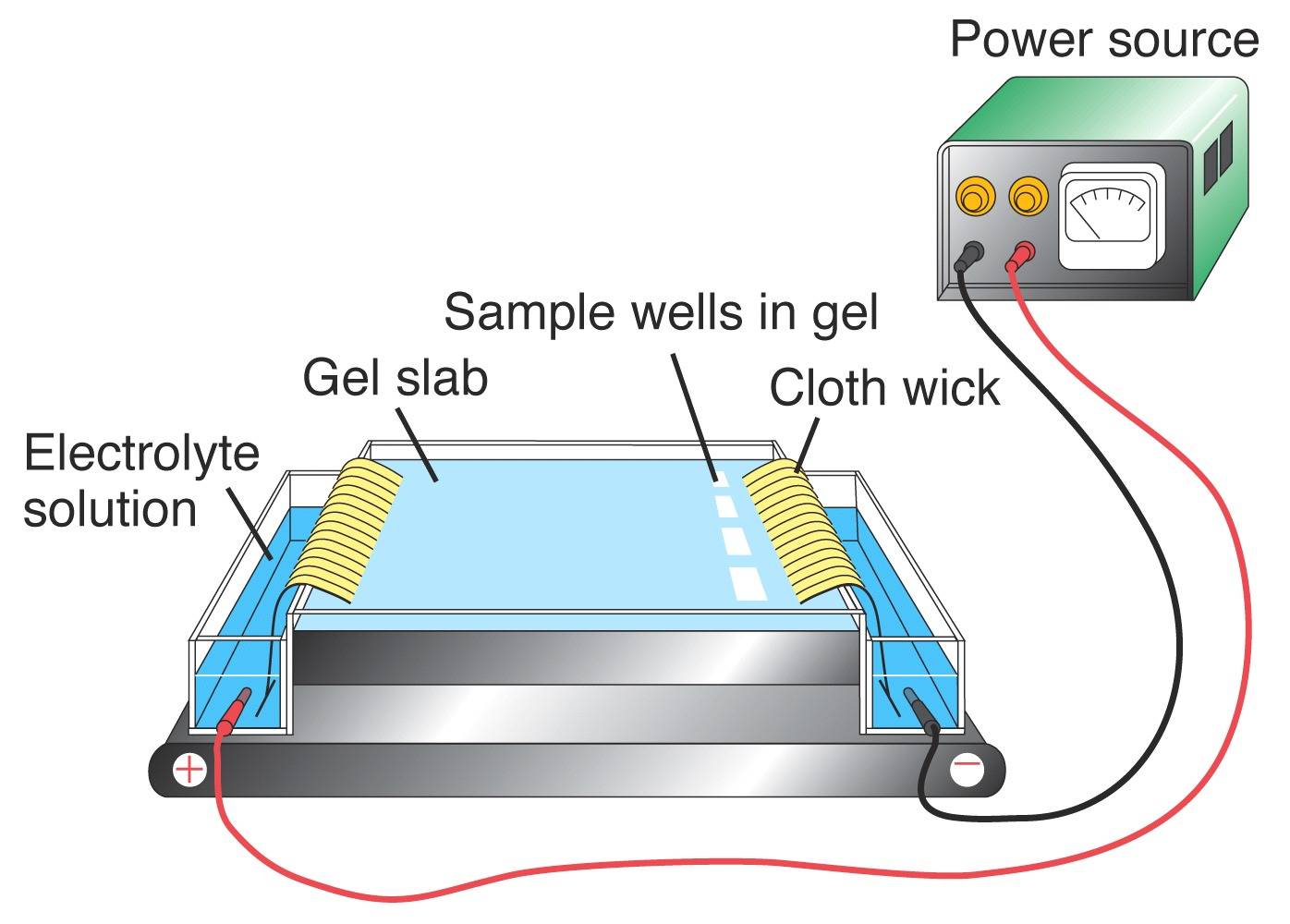
(左) 架空のアガロースゲル。 レーン1、2、3は、上記の個人のデータを使ったバンドを表しています。 レーン4は犯罪現場のDNAを表す。 (右) 典型的なアガロースゲル装置
臭化エチジウム(EtBr)は、DNAの塩基対に似た大きく平らな塩基性分子です。

臭化エチジウム
その化学構造のために、DNA鎖にインターカレート(挿入)することができます。 臭化エチジウムは、分子生物学の研究室で電気泳動ゲルの染色によく使われます。 この化合物は核酸と蛍光性の複合体を形成し、これをUV光で見ることができる。 EtBrはゲルを注入する前に温めたアガロースに最終濃度0.5ug/mlとなるように添加する。
Sequencing – Background
RFLP(リフリップと発音されることが多い)は、分子生物学者がDNAの特定の塩基配列が他の細胞に受け継がれるのを追跡するために使用する方法です。 RFLPは、さまざまな目的を達成するために、さまざまな場面で使用することができます。 RFLPは、父子関係や刑事事件において、DNAサンプルの出所を特定するために使用することができます。 また、RFLPは個人の病気の状態を判断するのにも使われます。
各生物は親からDNAを受け継いでいます。 DNAは世代ごとに複製されるので、どのような配列でも次の世代に引き継ぐことができます。 RFLPとは、DNAの両端に制限部位があり、その間に「標的」配列がある配列のことです。 ターゲット配列とは、相補的な塩基対を形成してプローブと結合できるDNAの断片のことです。 プローブとは、一本鎖DNAの配列に放射能や酵素のタグを付けて、プローブを検出できるようにしたものである。 プローブが標的と塩基対を形成すると、研究者はこの結合を検出することができ、プローブが検出可能であるため、標的配列がどこにあるかを知ることができる。 RFLPは、制限酵素とプローブ配列の特定の組み合わせでサザンブロットを行うと、一連のバンドを生成します。
例えば、制限酵素EcoR Iとターゲット配列20塩基 GCATGCATGCATGCATGCATで定義される特定のRFLPを追ってみましょう。

下に示したDNAのセグメントでは、RFLPの要素、つまり制限部位のペアに挟まれた標的配列を見ることができます。

2人の人間と、彼らが持っているこのRFLPを含むDNAの断片を見てみましょう(わかりやすくするために、2つのDNAのうちの1つだけを見ます)。 ジャックとジルはどちらも二倍体(二本鎖のDNA)の生物なので、このRFLPのコピーを2つ持っています。
- Jack 1: -GAATTC—(8.2 kb)—GCATGCATGCAT—(4.2 kb)—GAATTC-
- ジル1: -GAATTC—(8.2 kb)—GCATGCATGCATGCAT—(4.2 kb)—GAATTC-
このRFLPの2番目のコピーを調べてみると、それらは同一ではないことがわかります。
- Jack 2: -GAATTC—(1.8 kb)-CCCTTT–(1.2 kb)–GCATGCATGCATGCAT–(1.3 kb)-GAATTC-
- Jill 2: -GAATTC–(1.8 kb)-GAATTC–(1.2 kb)–GCATGCATGCATGCAT–(1.3 kb)-GAATTC-
従って、ジャックとジルが自分のDNAをRFLP分析にかけると、共通のバンドが1つと、分子量が相手と一致しないバンドが1つあることになります。

サザンブロット法
ここでは、サザンブロット(正式にはDNAブロットといいます)がどのように行われ、どのようなデータが得られるのかを簡単に説明します。

サザンブロットにより、研究者は制限断片の分子量を決定し、異なるサンプル中の相対量を測定することができます。
手順:
- DNA(ゲノムまたは他のソース)を制限酵素で消化し、ゲル電気泳動(通常はアガロースゲル)で分離します。 ゲル上には非常に多くの異なる制限断片が存在するため、通常は個別のバンドではなく塗抹標本のように見えます。
- DNAは、特殊なブロッティングペーパーのシートであるメンブレンに移されます。
- このブロットを、一本鎖のDNAであるプローブの多数のコピーと一緒にインキュベートします。 このプローブは、相補的なDNA配列と塩基対を形成して結合し、二本鎖DNA分子を形成します。
- プローブを無色の基質と一緒にインキュベートすることで、プローブの位置が明らかになります。プローブに結合した酵素は、目に見える色の生成物に変換するか、X線フィルムを露光する光を発します。
以下は、混合細胞集団に形質転換された遺伝子の存在を検出するために使用された実際のサザンブロットの例です。

左の図は、14種類のサンプルを載せた0.7%アガロースゲルの写真です(さらに右端のレーンには分子量マーカー、結果の分析には光る定規を使用しています)。 DNAの各サンプルは、同じ制限酵素(EcoRI)で消化されています。 DNAが一連の独立したバンドとしてではなく、スミアのように見えることに注目。
このゲルをレーザーでスキャンすると、以下のようなゲノム・フィンガープリントが得られます。