De DNA-vingerafdruk
Net als de vingerafdrukken die in de jaren dertig van de vorige eeuw in gebruik werden genomen door rechercheurs en politielaboratoria, heeft ieder mens een unieke DNA-vingerafdruk. In tegenstelling tot een conventionele vingerafdruk, die alleen op de vingertoppen voorkomt en door chirurgie kan worden gewijzigd, is een DNA-vingerafdruk voor elke cel, elk weefsel en elk orgaan van een persoon hetzelfde. Hij kan door geen enkele bekende behandeling worden gewijzigd. Bijgevolg wordt het nemen van DNA-vingerafdrukken snel de belangrijkste methode om individuele mensen te identificeren en van elkaar te onderscheiden. DNA-vingerafdrukken zijn ook nuttig voor verschillende toepassingen van onderzoek in de menselijke gezondheidszorg en voor de diagnose van erfelijke aandoeningen. DNA-vingerafdrukken worden vaak gebruikt in het rechtssysteem om verdachten te koppelen aan biologisch bewijsmateriaal en om vaderschapszaken op te lossen.
Hoe wordt DNA-vingerafdruk gebruikt om een misdadiger te identificeren?
Oxford Education (YouTube)
De polymerasekettingreactie
PCR zet de toon voor een wetenschappelijke revolutie
In 1983 ontwikkelde Kary Mullis bij Cetus Corporation een nieuwe techniek, de polymerasekettingreactie (PCR) genoemd. Het doel van PCR is om in een reageerbuis (in vitro) een grote hoeveelheid DNA te produceren, uitgaande van slechts een sporenhoeveelheid. Technisch gesproken betekent dit de gecontroleerde replicatie van een DNA-sequentie, of gen, van belang. De template-strengen kunnen elke vorm van dubbelstrengs DNA zijn, zoals genomisch DNA. Een onderzoeker kan sporen van genomisch DNA nemen uit een druppel bloed, een enkel haarzakje of een wangcel (in theorie is slechts één enkele sjabloonstreng nodig om miljoenen nieuwe identieke DNA-moleculen te kopiëren en te genereren) en voldoende maken om te bestuderen. Vóór PCR zou dit onmogelijk zijn geweest.
PCR-amplificatie
Geschat wordt dat het menselijk genoom 30.000-50.000 afzonderlijke genen bevat. De ware kracht van PCR is het vermogen om uit een volledig genoom miljoenen kopieën van een specifiek stuk DNA (of gen) te maken (of te amplificeren).
Het recept voor een PCR amplificatie van DNA bevat een eenvoudig mengsel van ingrediënten die herkenbaar zouden moeten zijn als de stukjes DNA die in het lichaam worden gebruikt om DNA te repliceren:
- DNA-sjabloon – bevat de intacte sequentie van het te amplificeren DNA
- Individuele deoxynucleotiden (A, T, G, en C) – de grondstof van DNA
- DNA-polymerase – een enzym dat de nucleotiden samenvoegt tot een nieuwe DNA-keten
- Magnesiumionen – een cofactor (katalysator) die DNA-polymerase nodig heeft om de DNA-keten te maken
- Oligonucleotide-primers – stukjes DNA die complementair zijn aan de template en die DNA-polymerase precies vertellen waar het moet beginnen met het maken van kopieën
- Zoutbuffer – zorgt voor het optimale ionische milieu en de optimale pH voor de PCR-reactie
Het template-DNA kan afkomstig zijn van bloed, huid, sperma of haar enz. Wanneer alle andere componenten onder de juiste omstandigheden worden gecombineerd, wordt een kopie gemaakt van het oorspronkelijke dubbelstrengs template-DNA-molecuul – waardoor het aantal template-strengen wordt verdubbeld. Telkens wanneer deze cyclus wordt herhaald, worden kopieën van kopieën gemaakt en verdubbelt het aantal template-strengen – van 2 naar 4 naar 8 naar 16 enzovoort – totdat er na 20 cycli 1.048.554 exacte kopieën van de doelsequentie zijn.
PCR maakt gebruik van dezelfde basisprocessen die cellen gebruiken om hun DNA te dupliceren.
- Complementaire DNA-streng hybridisatie
- DNA-strengsynthese via DNA-polymerase
De DNA-primers zijn zo ontworpen dat ze een DNA-sequentie in het genoom flankeren en zo het exacte startsignaal geven voor het DNA-polymerase om zich te binden en te beginnen met de synthese (replicatie) van kopieën van dat doel-DNA. Complementaire strenghybridisatie vindt plaats wanneer verschillende primers annealiseren, oftewel zich binden aan elk van hun respectieve complementaire basensequenties op het template-DNA. De primers zijn korte enkelstrengs DNA-moleculen (~20 basen lang), waarvan de ene complementair is aan een gedeelte van de 5′-3′-streng en de andere complementair is aan een gedeelte van de 3′-5′-streng van het sjabloon. Deze primers annealiseren aan de gescheiden template strengen en dienen als startpunten voor DNA Taq replicatie door DNA polymerase.
Taq DNA polymerase breidt de geannealde primers uit door de template streng te “lezen” en de complementaire sequentie te synthetiseren. Op deze wijze repliceert Taq polymerase de twee template DNA-strengen. Dit polymerase is geïsoleerd uit een hittestabiele bacterie (Thermus Aquaticus) die in de natuur leeft in de stoomopeningen in Yellowstone National Park. Daarom zijn de enzymen in deze bacterie geëvolueerd om hoge temperaturen (94°C) te weerstaan en kunnen ze in de PCR-reactie worden gebruikt.
PCR stap voor stap
PCR amplificatie omvat drie hoofdstappen, een denaturatiestap, een annealingstap, en een extensiestap (samengevat in de onderstaande figuur).
1) Denaturatie: Bij denaturatie wordt het reactiemengsel gedurende 1 minuut tot 94°C verhit, wat resulteert in het smelten of scheiden van het dubbelstrengs DNA-sjabloon in twee enkelstrengs moleculen.
2) Amplificatie: Bij PCR amplificatie moeten de DNA templates worden gescheiden voordat het polymerase een nieuwe kopie kan genereren. De hoge temperatuur die nodig is om de DNA-strengen te smelten zou normaal gesproken de activiteit van de meeste enzymen vernietigen, maar omdat Taq polymerase werd geïsoleerd uit bacteriën die gedijen in de hoge temperaturen van hete bronnen, blijft het actief.
3) Uitgloeien: Tijdens de annealing-stap “annealiseren” de primers van de oligonucleotide hun complementaire sequenties op de twee enkelstrengs DNA-sjabloonstrengen. In deze annealed posities kunnen zij fungeren als primers voor Taq DNA-polymerase. Zij worden primers genoemd omdat zij de synthese van een nieuwe streng “op gang brengen” door een korte sequentie dubbelstrengs DNA te leveren waaruit Taq polymerase zich kan uitbreiden om een nieuwe complementaire streng te vormen. De binding van de primers aan hun template-sequenties is ook sterk afhankelijk van de temperatuur.
Tijdens de extensie is het de taak van Taq DNA-polymerase om één voor één nucleotiden (A, T, G en C) aan de primer toe te voegen om zo een complementaire kopie van het DNA-sjabloon te maken.

Tijdens de polymerisatie bedraagt de reactietemperatuur 72°C, de temperatuur waarbij de activiteit van Taq polymerase optimaal is. De drie stappen van denaturatie, extensie en annealing vormen een “cyclus” van PCR. Een volledige PCR amplificatie ondergaat 40 cycli.
De volledige 40-cyclische reactie wordt uitgevoerd in een reageerbuis die in een thermocycler is geplaatst. De thermocycler bevat een aluminium blok waarin de monsters worden geplaatst en dat snel kan worden verwarmd en afgekoeld bij grote temperatuurverschillen. Het snel opwarmen en afkoelen van dit thermische blok staat bekend als temperatuurcycli of thermische cycli.
PCR temperatuurcyclus = denaturatiestap (94°C) + gloeistap (60°C) + verlengingsstap (72°C)
DNA Fingerprinting
Variable Number of Tandem Repeats (VNTR)
Het evolutionaire principe van variatie binnen een populatie is een hoeksteen in de biologie. Deze variatie is het gevolg van subtiele verschillen in de DNA-sequentie bij individuen van een bepaalde soort. Eén oorsprong van variatie ontstaat wanneer duplicatie van een kleine sequentie van nucleotiden optreedt tijdens DNA replicatie. Dit resulteert in een tandem-herhaling van de oorspronkelijke sequentie. Als deze fout bij een volgende replicatieronde opnieuw optreedt, komen er drie kopieën van een sequentie in tandem te staan, zoals hieronder wordt geschetst:
Individu 1: GTACTCCAATCATGTACCATGAC
Individu 2: GTACTCCAATCATCATGTACCATGAC
Individu 3: GTACTCATCATCATGTACCATGAC
Hier worden enkelvoudige DNA-strengen van dezelfde locus en drie verschillende individuen getoond. ‘CAT’ wordt één, twee of drie keer herhaald, wat resulteert in allelen van verschillende lengte.
Er zijn twee standaardmethoden voor DNA-fingerprinting:
- PCR van DNA dat VNTR’s bevat.
- Southern blotting (met behulp van RFLPs).
PCR maakt de amplificatie mogelijk van een enkele kopie van DNA tot miljoenen kopieën. Deze techniek vereist echter dat het DNA van belang reeds een bekende sequentie heeft om primers te kunnen ontwerpen die specifiek met het doel-DNA hybridiseren. Een dergelijk gebied dat in verschillende landen wordt gebruikt voor forensische analyse van DNA-monsters is de D1S80 locus. De D1S80 locus bevindt zich op het distale gedeelte van de korte arm van chromosoom 1 en bevat een variabel aantal tandem herhalingen.

D1S80 Voorbeeld Tandem Herhalingen
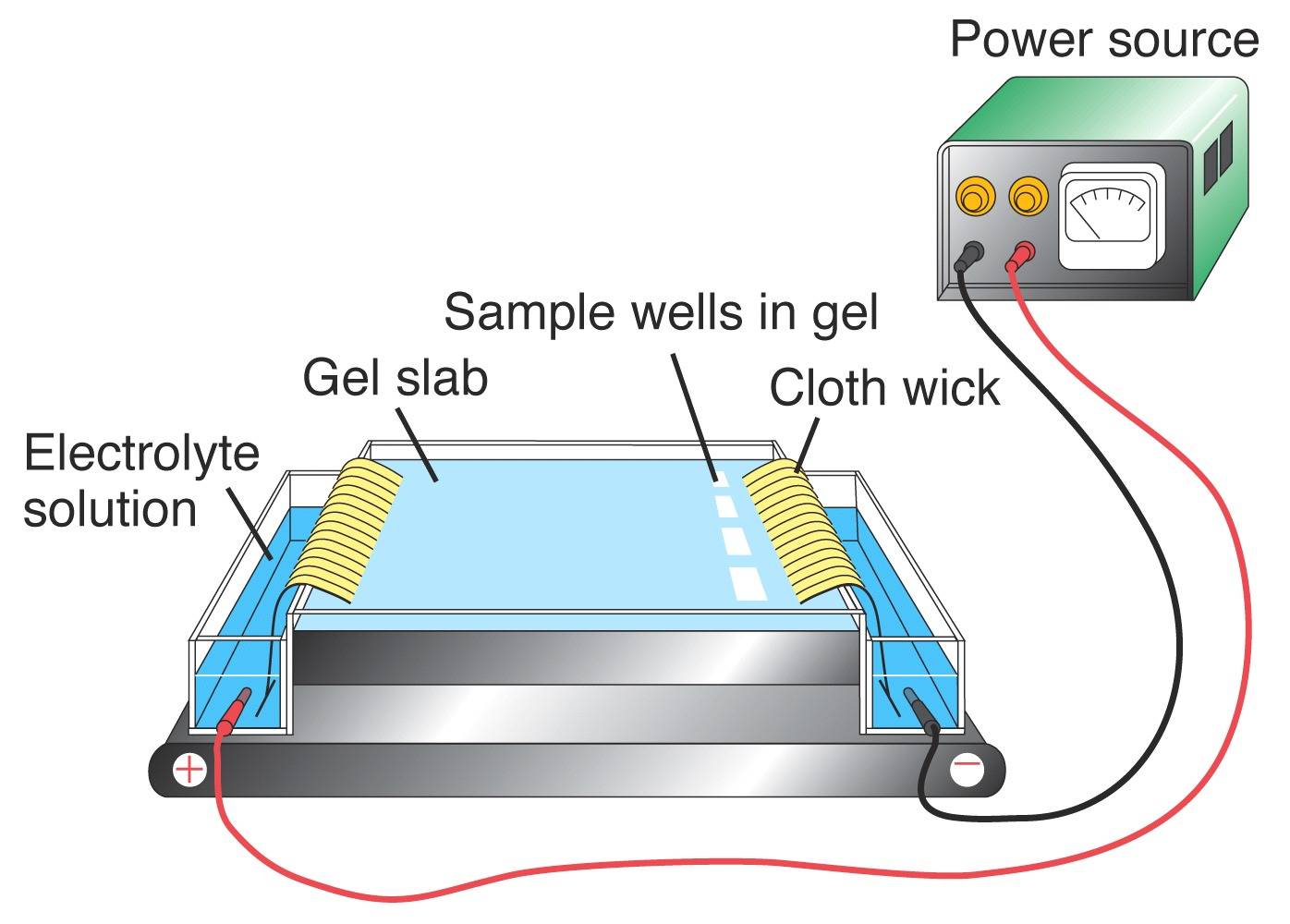
Electroforese
Met behulp van PCR kunnen delen van DNA waarvan bekend is dat ze VNTR’s bevatten, worden geamplificeerd. Het resulterende product wordt vervolgens bekeken om het individuele DNA-genotype te verkrijgen. Om het DNA-profiel van een bepaalde locus zichtbaar te maken, moeten de DNA-fragmenten met behulp van gelelektroforese op basis van hun grootte worden gescheiden. Er wordt een elektrische stroom aangelegd op een agarosegelmatrix. Aangezien DNA negatief geladen is door de fosfaatgroepen, zullen de fragmenten naar de positieve pool bewegen. DNA-moleculen van verschillende grootte migreren door de gel met verschillende snelheden, waarbij de kleinere fragmenten sneller bewegen dan de grotere. Zodra het DNA in de gel is gekleurd, kan het worden gevisualiseerd om het DNA-profiel te onthullen.

(Links) Fictieve agarosegel. De rijstroken 1, 2 en 3 geven de banden weer met de gegevens van de hierboven genoemde personen. Baan 4 vertegenwoordigt DNA van een plaats delict. (Rechts) Typisch agarosegel-apparaat.
Ethidiumbromide (EtBr) is een groot, plat basisch molecuul dat lijkt op een DNA-basenpaar.

Ethidiumbromide
Omwille van zijn chemische structuur kan het in een DNA-streng intercaleren (of invoegen). Ethidiumbromide wordt algemeen gebruikt in moleculair-biologische laboratoria om elektroforese-gels te kleuren. De verbinding vormt fluorescerende complexen met nucleïnezuren en deze kunnen onder UV-licht worden bekeken. EtBr wordt vóór het gieten van de gel aan de verwarmde agarose toegevoegd tot een eindconcentratie van 0,5 mg/ml. EtBr is een mutageen en moet met uiterste voorzichtigheid en met handschoenen aan worden gehanteerd.
Sequencing – Achtergrond
RFLP (vaak uitgesproken als “rif lip”, alsof het een woord is) is een methode die door moleculaire biologen wordt gebruikt om een bepaalde sequentie van DNA te volgen terwijl het aan andere cellen wordt doorgegeven. RFLP’s kunnen in veel verschillende situaties worden gebruikt om verschillende doelstellingen te bereiken. RFLP’s kunnen worden gebruikt in vaderschapszaken of strafzaken om de bron van een DNA-monster te bepalen. RFLP’s kunnen ook worden gebruikt om de ziektestatus van een individu te bepalen.
Elk organisme erft zijn DNA van zijn ouders. Aangezien DNA bij elke generatie wordt gerepliceerd, kan een bepaalde sequentie aan de volgende generatie worden doorgegeven. Een RFLP is een sequentie van DNA die aan elk uiteinde een restrictieplaats heeft met daartussen een “doel”-sequentie. Een doelsequentie is elk DNA-segment dat zich aan een probe kan binden door complementaire basenparen te vormen. Een probe is een sequentie van enkelstrengs DNA die is gemerkt met radioactiviteit of een enzym, zodat de probe kan worden gedetecteerd. Wanneer een probe basenparen vormt met zijn doelwit, kan de onderzoeker deze binding detecteren en weten waar de doelsequentie zich bevindt, aangezien de probe detecteerbaar is. Een RFLP levert een reeks banden op wanneer een Southern blot wordt uitgevoerd met een bepaalde combinatie van restrictie-enzym en probe-sequentie.
Hier volgen we bijvoorbeeld een bepaalde RFLP die wordt gedefinieerd door het restrictie-enzym EcoR I en de doelsequentie van 20 basen GCATGCATGCATGCATGCAT. EcoR I bindt aan zijn herkenningssequentie GAATTC en knipt het dubbelstrengs DNA door zoals te zien is:

In het hieronder getoonde DNA-segment zie je de elementen van een RFLP; een doelsequentie geflankeerd door een paar restrictieplaatsen. Wanneer dit DNA-segment door EcoR I wordt doorgesneden, ontstaan er drie restrictiefragmenten, maar slechts één daarvan bevat de doelsequentie die door de complementaire probe-sequentie (paars) kan worden gebonden.

Laten we eens kijken naar twee mensen en de DNA-segmenten die zij bij zich dragen en die deze RFLP bevatten (voor de duidelijkheid zien we slechts een van de twee DNA-segmenten). Aangezien Jack en Jill beide diploïde (dubbelstrengs DNA) organismen zijn, hebben zij twee kopieën van deze RFLP. Wanneer we één exemplaar van Jack en één exemplaar van Jill onderzoeken, zien we dat ze identiek zijn:
- Jack 1: -GAATTC—(8,2 kb)—GCATGCATGCATGCATGCAT—(4.2 kb)—GAATTC-
- Jill 1: -GAATTC–(8,2 kb)—GCATGCATGCAT–(4,2 kb)—GAATTC-
Als we hun tweede exemplaren van deze RFLP bekijken, zien we dat ze niet identiek zijn. Jack 2 mist een EcoR I restrictie site die Jill 1.2 kb stroomopwaarts van de doelsequentie heeft (verschil cursief).
- Jack 2: -GAATTC–(1.8 kb)-CCCTTT–(1.2 kb)–GCATGCATGCATGCAT–(1.3 kb)-GAATTC-
- Jill 2: -GAATTC–(1.8 kb)-GAATTC–(1.2 kb)–GCATGCATGCATGCAT–(1.3 kb)-GAATTC-
Dus, wanneer Jack en Jill hun DNA aan een RFLP-analyse onderwerpen, zullen zij één band gemeen hebben en één band die in molecuulgewicht niet overeenkomt met die van de ander:

Southern Blot Methode
Dit is een kort overzicht van hoe een Southern blot (meer formeel een DNA-blot genoemd) wordt uitgevoerd en wat voor soort gegevens je er uit kunt halen. Het resultaat van een Southern Blot is waar de meeste mensen aan denken als ze het hebben over een “DNA-vingerafdruk”.

Southern blots stellen onderzoekers in staat het moleculaire gewicht van een restrictiefragment te bepalen en relatieve hoeveelheden in verschillende monsters te meten.
Procedure:
- DNA (genoom of een andere bron) wordt met een restrictie-enzym gedigesteerd en door gelelektroforese gescheiden, meestal een agarosegel. Omdat er zoveel verschillende restrictiefragmenten op de gel voorkomen, ziet het er meestal eerder als een uitstrijkje uit dan als afzonderlijke banden. Het DNA wordt tot afzonderlijke strengen gedenatureerd door incubatie met NaOH.
- Het DNA wordt overgebracht op een membraan, een vel speciaal vloeipapier. De DNA-fragmenten behouden hetzelfde scheidingspatroon dat ze op de gel hadden.
- De blot wordt geïncubeerd met vele kopieën van een probe die uit enkelstrengs DNA bestaat. Deze probe zal basenparen vormen met zijn complementaire DNA-sequentie en zich binden om een dubbelstrengs DNA-molecuul te vormen. De probe kan niet worden gezien, maar is radioactief of er is een enzym aan gebonden (b.v. alkalische fosfatase of mierikswortelperoxidase).
- De plaats van de probe wordt zichtbaar gemaakt door deze te incuberen met een kleurloos substraat dat door het toegevoegde enzym wordt omgezet in een gekleurd product dat kan worden gezien of licht afgeeft dat röntgenfilm belicht. Als de probe met radioactiviteit is gelabeld, kan hij direct röntgenfilm belichten.
Hieronder ziet u een voorbeeld van een echte Southern blot die wordt gebruikt om de aanwezigheid van een gen op te sporen dat in een gemengde celpopulatie is getransformeerd. In deze Southern blot is gemakkelijk vast te stellen welke cellen het gen hebben geïncorporeerd en welke niet.

De figuur links toont een foto van een 0,7% agarose-gel waarop 14 verschillende monsters zijn geladen (plus een molecuulgewichtmarker in de uiterst rechtse lane en een oplichtende liniaal die voor de analyse van de resultaten wordt gebruikt). Elk DNA-monster is gedigesteerd met hetzelfde restrictie-enzym (EcoRI). Merk op dat het DNA niet verschijnt als een reeks discrete banden, maar eerder als een uitstrijkje. De figuur rechts is een kopie van de röntgenfilm en laat zien welke stammen het doel-DNA bevatten en welke niet.
Een laserscan van de gel levert een genomische vingerafdruk op van het onderstaande type:
